Vol 8 No 4 2023 – 29
Enny Ganchozo-Mendoza1, Francisco J. Flores 2, 3; Felipe R. Garcés-Fiallos4*
1 Instituto de Posgrado, Maestría en Sanidad Vegetal, Universidad Técnica de Manabí. Código Postal 130105, Portoviejo, Manabí, Ecuador.
2 Departamento de Ciencias de la Vida y la Agricultura, Universidad de las Fuerzas Armadas ESPE, Sangolquí 170501, Ecuador.
3 Centro de Investigación de Alimentos, CIAL, Facultad de Ciencias de la Ingeniería e Industrias, Universidad UTE, Quito, Ecuador.
4 Laboratorio de Fitopatología, Campus Experimental La Teodomira, Facultad de Ingeniería Agronómica, Universidad Técnica de Manabí, Santa Ana EC 131301, Ecuador.
*Corresponding author: Email: felipe.garces@utm.edu.ec Mobile: +593 939929958
Available from: http://dx.doi.org/10.21931/RB/2023.08.04.30
RESUMEN
El tabaco (Nicotiana tabacum L.) es un cultivo industrial de importancia económica y una planta modelo importante, cultivada ampliamente en muchos países. Su producción, rendimiento y calidad se han visto gravemente afectados por una serie de factores entre las que destacan las infecciones virales. Hasta el momento se han reportado más de 60 virus pertenecientes a 20 géneros que infectan y provocan pérdidas sustanciales de rendimiento en tabaco. Los genomas de estos fitopatógenos pueden ser de ADN o ARN, distribuyéndose en varias partículas virales (monopartito, bipartito o tripartito). En su gran mayoría, las infecciones virales en tabaco son ocasionadas por virus de ARN monocatenarios de sentido positivo (ARN +) como los pertenecientes al género Tobamovirus y Cucumovirus. No obstante, los Begomovirus (virus de ADN) también impactan económicamente el cultivo de tabaco. Esta revisión enlista los principales virus de ADN y ARN que infectan plantas de tabaco, así como sus vectores más relevantes. Además, se abordan las técnicas de detección y diagnóstico que se han desarrollado continuamente para identificar correctamente las enfermedades virales asociadas al cultivo de tabaco.
Palabras clave: Nicotiana tabacum L., virus de plantas, incidencia de virus, detección y diagnóstico viral
ABSTRACT
Tobacco (Nicotiana tabacum L.) is an economically significant industrial crop and an important model plant, widely grown in many countries. Its production, yield and quality have been severely impacted by a series of factors, with viral infections being prominent among them. To date, more than 60 viruses from 20 genera have been reported to infect and cause substantial yield losses in tobacco. The genomes of these phytopathogens can be either DNA or RNA, distributed in various viral particles (monopartite, bi-partite or tripartite). Predominantly, viral infections in tobacco are caused by single-stranded positive-sense RNA viruses (+RNA), such as those belonging to the Tobamovirus and Cucumovirus genera. However, Begomoviruses (DNA viruses) also economically impact the tobacco crops. This review lists the major DNA and RNA viruses infecting tobacco plants, along with their most relevant vectors. Additionally, it outlines the continuously developed detection and diagnostic techniques continuously essential for accurately identifying viral diseases associated with tobacco cultivation.
Key words: Nicotiana tabacum L., plant viruses, virus incidence, virus detection and diagnosis.
INTRODUCCION
El tabaco (Nicotiana tabacum L.) es una especie endémica de América del Sur perteneciente a la familia Solanaceae, cultivada en todo el mundo 1. Desde el punto de vista económico, es la especie más importante del género Nicotiana, existiendo aproximadamente 152 variedades cultivadas 2,3 Esta solanácea es atacada por un sinnúmero de fitopatógenos que afectan principalmente el área foliar, destacándose los virus. Estos agentes infecciosos son económicamente importantes y el manejo de sus enfermedades es crucial para la producción agrícola 4–6.
Un gran número de virus pueden infectar plantas de tabaco ya sea de forma natural o experimental. Estos agentes infecciosos están distribuidos mundialmente, reduciendo el rendimiento y calidad del cultivo, representando un factor económico importante para los tabacaleros 4–6. Los síntomas característicos asociados a virosis en tabaco son muy variados i.e. manchas anulares cloróticas y necróticas, mosaico, enanismo, moteado, estriado, arrugamiento, deformaciones, entre otros 7–10. Es esencial realizar una identificación temprana de los virus en las plantas de tabaco con el fin de minimizar los daños y pérdidas asociadas.
Entre los virus más importantes del tabaco se incluyen a Tobamovirus (como Tobacco mosaic virus – TMV) que es considerado uno de los agentes virales de mayor importancia económica, seguido de Cucumovirus (Cucumber mosaic virus – CMV), Potyvirus (Potato virus Y – PVY; Tobacco vein mottling virus – TVMV y Tobacco etch virus -TEV), Begomovirus (Tobacco leaf curl virus -TLCV), y Tospovirus (Tomato spotted wilt orthotospovirus – TSWV), que afectan la producción y el rendimiento de las hojas de tabaco en muchas plantaciones a nivel mundial 11–15. Dado que los síntomas de las virosis se manifiestan en gran medida en las hojas, que constituyen el producto principal del cultivo de tabaco, resulta crucial recopilar y sintetizar el conocimiento disponible acerca de estas enfermedades virales. En este sentido, se ha llevado a cabo una revisión actualizada de los virus que afectan al cultivo de tabaco, su incidencia y daños producidos, su relación con los vectores más relevantes, así como las técnicas actuales para su detección e identificación.
Virus que afectan plantas de tabaco
El tabaco es una de las solanáceas más susceptibles a las infecciones virales, con más de 20 géneros de virus con capacidad de infectarlo de forma natural. Además, se ha demostrado experimentalmente que las plantas de tabaco pueden ser infectadas por más de un centenar de especies virales 16. Se han documentado casos de infecciones mixtas en las que coexisten dentro del mismo hospedero más de una especie o variantes genéticamente distintas de la misma especie. Los virus involucrados pueden interactuar entre sí, dando lugar a fenómenos de sinergismo, neutralismo o antagonismo 17,18.
Los virus pueden presentar genomas de ADN o ARN, los cuales pueden estar contenidos en una sola o distribuidos en varias partículas virales (monopartito, bipartito o tripartito) 19. Las infecciones en plantas de tabaco en su mayoría son ocasionadas por virus de ARN monocatenarios de sentido positivo (ARN +) 20. No obstante, hay un grupo importante de virus de ADN como los Begomovirus, que causan un impacto económico al cultivo. En la Tabla 1 se presentan algunas de las especies virales que infectan plantas de tabaco, así como sus vectores asociados.
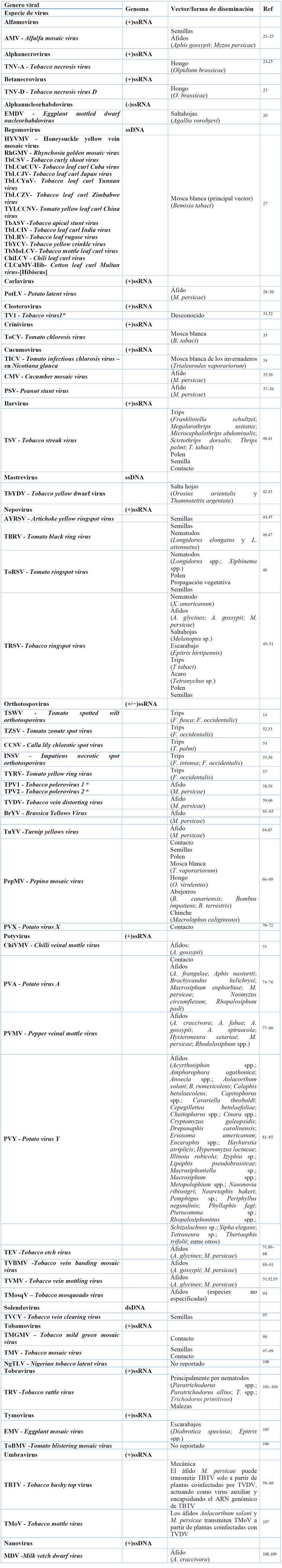
* Nombre científico tentativo
Tabla 1. Virus y sus vectores identificados y reportados en plantas de tabaco (Nicotiana tabacum L.)
Principales síntomas asociados a virus en tabaco
Las infecciones por virus pueden causar diversos síntomas en las plantas de tabaco, como atrofia, patrones de mosaico, amarillamiento, hojas enrolladas, manchas anulares, necrosis, marchitamiento y otras anomalías durante su desarrollo 110. TMV es uno de los virus más comunes e importantes en N. tabacum, presentando diferentes síntomas i.e. clorosis, oscurecimiento de nervaduras en hojas, moteado, manchas, mosaicos y retraso en el crecimiento 111–114. Por otro lado, plantas de tabaco infectadas con el Begomovirus CMV generalmente muestran clorosis, mosaicos, necrosis, enanismo, retraso en el crecimiento y malformación foliar. La expresión cualitativa de estos síntomas en plantas de tabaco depende de la cepa de CMV 115–117. Algo parecido sucede con los síntomas presentados por TEV, asociados principalmente a clorosis, deformación y reducción del crecimiento 118.
Otros virus como el Potyvirus PVY induce síntomas en plantas de tabaco que también dependen del tipo de cepa viral, como necrosis en las venas de hojas, clorosis, ocasionalmente también una distorsión foliar y necrosis en tallos asociados a PVYN, y moteado y mosaico causados por PVYO 119–121. PVA, es otro Potyvirus que afecta tabaco y causa moteado difuso muy leve y aclaramiento de venas en hojas 74. Finalmente, Alfamovirus como AMV induce en plantas de tabaco puntos cloróticos y manchas en hojas, pudiendo estos tejidos presentar venas pálidas, anillos blanquecinos y patrones de líneas de tejido necrótico, y deformación, e incluso a veces mostrar un patrón de mosaico brillante 122.
Las infecciones mixtas de dos o más virus no relacionados es un fenómeno frecuente en la naturaleza. Esta sinergia a menudo se manifiesta por un aumento notable tanto en la expresión de síntomas como en la acumulación de virus, en comparación con una sola infección. Una de estas interacciones es la de PVY con el Potexvirus PVX en tabaco, la cual puede producir desde amarillamiento en hojas hasta una necrosis severa 123,124. Otro ejemplo, es la coinfección viral entre TMV y CMV; la aparición de síntomas es más temprana y grave que la monoinfección en plantas de tabaco 125. En la Figura 1 se pueden observar algunos de los síntomas virales encontrados en plantas comerciales de tabaco.

Figura 1. Plantas de tabaco con síntomas virales aparentes: (A) Mosaico y malformaciones en hojas jóvenes, (B) aclaramiento de nervaduras en hojas y reducción foliar, y (C) patrón de mosaico y reducción de crecimiento en hojas jóvenes.
Transmisión de virus en plantas de tabaco
El tabaco al igual que la mayoría de las plantas, adquieren virus a través de vectores que transmiten las partículas virales desde una planta huésped infectada. Generalmente, estos vectores son artrópodos, insectos perforadores-chupadores fitófagos (pulgones, salta hojas, moscas blancas, trips, etc.), pero también escarabajos, ácaros, nematodos, plantas parásitas, malezas, hongos y protistas. Sin embargo, la transmisión viral en tabaco también puede ocurrir por contacto mecánico, semilla botánica (sexual) y polen 99,126–131. Los vectores pueden transmitir virus de manera persistente, semipersitente y no persistente. Esta clasificación está dada por la localización del virus en el vector, tiempo requerido por el vector para la adquisición del virus, retención y transmisión del virus, y asociación del virus con varios órganos internos del vector 92,131. Además, se han identificado proteínas codificadas por diferentes virus de plantas para interactuar específicamente con sus respectivos vectores (generalmente insectos) para facilitar la transmisión de virus 132.
En el modo no persistente, los insectos vectores retienen los virus en sus estiletes. En el modo semipersistente, los virus se transportan a los intestinos anteriores o a las glándulas salivales del vector, pero no pueden propagarse de los intestinos a las glándulas salivales o ingresar a la hemolinfa. En el modo persistente, los virus se retienen en los intestinos del vector y pueden propagarse a las glándulas salivales. En los modos no persistente y semipersistente, los virus se retienen por un corto tiempo. Contrariamente, en el modo persistente los virus son retenidos por un tiempo relativamente largo y se los pueden encontrar en la hemolinfa de los vectores 128.
Los insectos, especialmente áfidos y moscas blancas son los principales vectores de virus que infectan plantas de tabaco, representando más del 50% de todos los transmisores de fitovirus 133. Estos vectores utilizan diversos mecanismos de transmisión, por ejemplo, los Begomovirus son transmitidos de manera persistente por moscas blancas, mientras que los Closterovirus son transmitidos por áfidos de forma semipersistente. Aunque otros virus como los Potyvirus cuentan con numerosas especies transmitidas de forma no persistente por pulgones, estos también pueden ser trasmitidos por semillas e inoculación mecánica. Por otra parte, los Tobamovirus no se transmiten a través de vectores, pero pueden propagarse mediante el contacto entre plantas y, en ocasiones, a través de semillas 19,127.
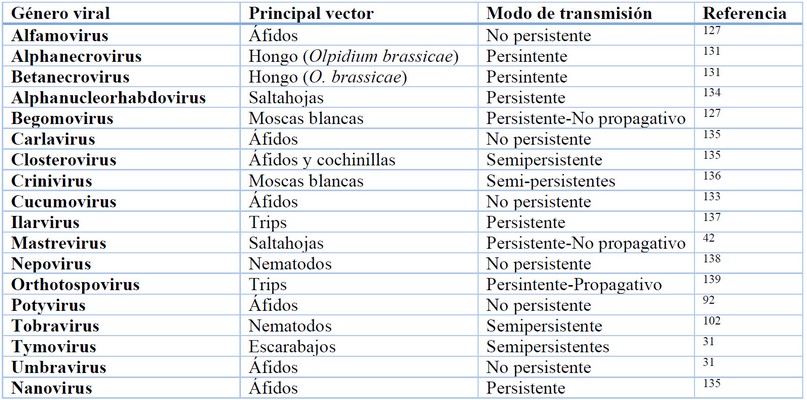
Tabla 2. Géneros virales que infectan plantas de tabaco (Nicotiana tabacum L.) y son transmitidos por vectores y de otras maneras
Incidencia viral en plantas de tabaco
Se han realizado varios estudios sobre la incidencia y distribución de virus en plantaciones de tabaco como primer paso hacia la búsqueda de estrategias de manejo. En Turquía, mediante el método serológico DAS-ELISA (double-antibody sandwich enzyme-linked immunoabsorbent assay) es encontró una tasa de infección del 61,5% para TSWV 140 en hojas de tabaco. En el mismo país, se han reportado incidencias de 8,8, 5,5 y 0,2% correspondientes a CMV, TMV e infecciones mixtas TMV + CMV, respectivamente 141. En Asia, específicamente en China, usando técnicas moleculares encontraron una incidencia entre 0,5 y 3% para ChiVMV en muestras provenientes de un cultivo comercial de tabaco 142.
Es importante destacar la importancia que tiene el uso de métodos de detección o identificación de virus en plantas de tabaco. Por ejemplo, en Irán se encontró una incidencia de 79% atribuida inicialmente a la infección con TSV con base en la sintomatología observada en diferentes cultivares. Sin embargo, los análisis moleculares subsiguientes revelaron que prácticamente ninguna de las muestras albergaba una única infección de TSV. Más bien, se constató que estaban afectadas por coinfecciones de uno o varios virus, entre ellos TSWV, CMV, PVY y TMV 143. En Serbia, de un total de 26 muestras foliares de tabaco comercial y analizadas mediante técnicas serológicas y moleculares, se encontró que 25 fueron positivas para TSWV 144. La incidencia de las infecciones virales depende de diferentes factores como del cultivar usado, la edad de las plantas, prácticas de manejo, entre otros componentes. Al respecto, en varias áreas geográficas de Grecia se estudiaron tres cultivares de tabaco, encontrándose una incidencia viral entre 0,4 y 80 % en plántulas establecidas en semilleros de hasta el 100 % en plantas en condiciones de campo 145.
Aunque el uso de técnicas serológicas o moleculares son importantes en la identificación de virus en plantas de tabaco, la cuantificación de virosis en condiciones de campo también es muy importante. En El Empalme, Ecuador, evaluando la presencia de virosis en plantas comerciales de tabaco cultivares Connecticut y Sumatra durante los años 2021 y 2022, se encontraron síntomas aparentes i.e. mosaicos, enanismo, reducción y distorsión de la lámina foliar, necrosis, clorosis, y aclaramiento de nervaduras (Figura 2). Durante las dos campañas en esta localidad, los síntomas virales se observaron a partir de los 15 días después del trasplante (ddt). La mayor incidencia (4% en promedio) en ambos cultivares de tabaco y campañas fueron a los 28 ddt. A los 42 ddt, la incidencia de virosis comienza a disminuir coincidiendo con la floración en ambos cultivares. Cabe destacar que las plantas determinadas como infectadas se eliminaron de la parcela. Resultados como estos pueden considerarse una amenaza importante para cualquier país, teniendo en cuenta que las parcelas usadas en ese estudio fueron manejadas comercialmente, es decir, se aplicaron insecticidas, se desinfectaron herramientas y se eliminaron plantas con síntomas virales aparentes.

Figura 2. Incidencia de virosis en plantas comerciales de tabaco cultivares Connecticut y Sumatra durante los años 2021 (A) y 2022 (B). Las barras en cada tiempo de evaluación representan la desviación estándar. El Empalme, Guayas, Ecuador.
Identificación y caracterización de virus fitopatógenos
Contrariamente a la mayoría de fitopatógenos, los virus son parásitos obligados, dificultando su identificación y caracterización. No obstante, a través del tiempo se han desarrollado diferentes técnicas basadas en el fenotipo de los virus, incluyendo, pruebas de transmisión, técnicas bioquímicas, microscopía electrónica, y técnicas relacionadas con las proteínas de la cápside viral como ELISA (ensayo inmunoabsorbente ligado a enzimas), microscopía inmunoelectrónica, inmunotransferencia e inmunosensores de microbalanza de cristal de cuarzo. También se han desarrollado métodos basados en el análisis de ácidos nucleicos como ensayos de hibridación, reacción en cadena de la polimerasa (PCR) y sus variantes (RT-PCR- Real Time, entre otras) 146.
Las técnicas serológicas de ELISA y moleculares como hibridación molecular y amplificación de ADN son rápidas y específicas para la detección de fitovirus. ELISA se basa en la unión específica de proteínas virales con anticuerpos, mientras que la de hibridación molecular en la unión de ácidos nucleicos virales con sondas de ADN o ARN de secuencia específica. Estos sucesos de unión se visualizan mediante marcadores basados en colorantes fluorescentes, enzimas que producen reacciones colorimétricas o quimioluminiscentes 147. El uso de cualquiera de estas técnicas es importante en la identificación de fitovirus. Por ejemplo, entre 1997 y 2000, en las principales regiones productoras de tabaco de Grecia, el empleo de la técnica ELISA permitiendo detectar algunos virus como AMV, CMV, EMDV, PVY, TMV y TSWV 145.
Los métodos de detección basados en la amplificación de ácidos nucleicos más utilizados para la detección de patógenos son la reacción en cadena de polimerasa (PCR) y las amplificaciones isotérmicas. La PCR es una reacción in vitro mediada por cebadores moleculares para amplificar secuencias de ácidos nucleicos diana, basándose en la especificidad de la hibridación y replicación del ADN o ADN complementario (ADNc) en el caso de los virus de ARN 148,149. Una PCR estándar es un procedimiento de tres pasos: (1) desnaturalización a alta temperatura (90–95 ºC), (2) hibridación de cebadores específicos del objetivo (45–60 ºC), y (3) extensión del cebador mediante una ADN polimerasa termoestable a 72 ºC, obteniendo así millones de copias de ácidos nucleicos objetivo 150.
Se han desarrollado diferentes variantes de PCR para mejorar la sensibilidad y especificidad para la detección de fitovirus, reportándose entre estos a la PCR anidada (nPCR), PCR de inmunocaptura (IC-PCR), PCR multiplex (M-PCR), PCR en tiempo real, y huella digital de ADN. También, están la PCR acoplada con la transcripción inversa de ARN (RT-PCR), RT-PCR en tiempo real (RT-qPCR), RT-PCR con inmunocaptura (IC-RT-PCR), y AmpliDet RNA. Todos estos métodos permiten una detección y cuantificación rápida y precisa de virus en plantas 151–153. Estudios realizados para la detección de cinco especies de Tobamovirus serológicamente relacionados en N. tabacum y N. benthamiana demuestran que tanto la RT-PCR-RFLP (usando cebadores con alta homología) como la RT-PCR (usando cebadores específicos de especie) son adecuados para detectar infecciones mixtas y útiles para su diagnóstico 154. De la misma manera, el uso de la RT-PCR en multiplex, ha permitido la detección simultánea de TBTV, TVDV, satellite RNA TBTV -Sat-TBTV en plantas de tabaco 155.
En los últimos años se vienen utilizando otras técnicas como la amplificación isotérmica, que se puede lograr mediante dos enfoques: 1) amplificación dependiente de helicasa (HAD): utiliza una HAD para separar las hebras de dsDNA que permite la unión del cebador y la extensión mediante la ADN polimerasa a una temperatura constante aproximada de 65 °C 156, 2) amplificación mediante polimerasa y recombinasa (RPA): utiliza una recombinasa, proteínas de unión a ADN monocatenario y ADN polimerasa de desplazamiento de cadena a una temperatura entre 37 y 42 °C 157. Otro método isotérmico es la amplificación basada en secuencias de ácidos nucleicos (NASBA). Esta herramienta consta de una transcriptasa inversa para producir ADN a partir de las plantillas de ARN, la ARN polimerasa T7, la ARNasa H y dos cebadores de oligonucleótidos 151.
También pertenece a las técnicas isotérmicas, la amplificación de círculo rodante (RCA) que requiere un pequeño ADN circular monocatenario como plantilla para generar moléculas largas de ADN o ARN monocatenarias con múltiples unidades repetitivas que corresponden a la plantilla de ADN circular a 37 °C 158. Esta técnica se ha empleado en China para la detección del Nanovirus MVDV en plantas de tabaco 7.
Uno de los métodos isotérmicos más empleados es la amplificación isotérmica mediada por bucle (LAMP), que se basa en el autociclado y la alta actividad de desplazamiento de la cadena de ADN mediada por la polimerasa Bst de Geobacillus stearothermophilus, en condiciones isotérmicas de 60 a 65 °C. Esta técnica proporciona una alta especificidad debido al uso de cuatro a seis cebadores que reconocen entre seis y ocho regiones independientes, todas ellas dirigidas a una región diana específica 159. Se han implementado algunas variantes de LAMP, incluida la amplificación isotérmica mediada por bucle de transcripción inversa (RT-LAMP), que utiliza transcriptasa inversa combinada con ADN polimerasa para detectar secuencias de ARN 160,161. La técnica RT-LAMP ha demostrado ser extremadamente útil en el diagnóstico de muchos virus de ARN.
Recientemente, se han implementado nuevos métodos para el diagnóstico molecular de fitovirus. Uno de ellos es el basado en el sistema de inmunidad procariótico de repeticiones palindrómicas cortas agrupadas regularmente interespaciadas (CRISPR)/genes asociados a CRISPR (Cas), que es un sistema inmunitario adaptativo que ha sido reprogramado en una tecnología de selección de genes precisa, simple y eficiente 162. Esta novel tecnología ha revolucionado diversas áreas de las ciencias de la vida, la medicina y la biotecnología, donde se incluyen a la fitopatología. Aunque está técnica molecular está siendo usada intensamente en el fitomejoramiento y desarrollo de germoplasma con rasgos beneficiosos mediante el uso de vectores virales para introducir los reactivos CRISPR/Cas en las células vegetales, también se la ha empleado en el diagnóstico molecular de fitovirus163. Por ejemplo, utilizando el sistema CRISPR-Cas12a sensible y específico se han podido detectar en N. benthamiana los Geminivirus TYLCV y ToLCNDV 164.
La metagenómica es otra de las técnicas empleadas en la identificación de fitovirus, mediante el uso de la secuenciación de alto rendimiento. En este método, se extrae el ácido nucleico total de una muestra infectada, que incluye tanto al huésped como los patógenos virales, y luego se realiza la secuenciación de las secuencias utilizando herramientas bioinformáticas para identificar los virus presentes 165. Una de la ventajas que presenta la técnica, es que esta secuenciación de alto rendimiento permite la identificación y caracterización del genoma completo de un virus sin necesidad de un conocimiento previo sobre su presencia en la muestra 166. Este método utiliza plataformas que realizan secuenciación masiva y paralela, lo que significa que millones de fragmentos de ácidos nucleicos de una sola muestra se secuencian simultáneamente.
La aplicación de HTS en virología vegetal abarca la secuenciación, descubrimiento y detección de virus/viroides, así como estudios de ecología, epidemiología, replicación y transcripción viral. Es considerada una técnica sensible, precisa y rápida 167,168. No obstante, los datos generados por la HTS se convierten en grandes conjuntos de datos sin procesar, conocidos como «big data», que requieren un análisis adicional para extraer información relevante. En el análisis de secuencias, se utilizan pipelines (flujos de trabajo) de software específicos para cada etapa, siendo importante analizar la calidad de las lecturas obtenidas.
Cuando se trabaja con HTS, es importante realizar una limpieza bioinformática para eliminar lecturas y fragmentos de baja calidad. Una vez que se tienen las lecturas limpias, se lleva a cabo el ensamblaje. Este proceso implica encontrar superposiciones entre las lecturas para reconstruir secuencias de consenso o cóntigos (contigs) más largos. En la etapa final del análisis, se comparan los contigs obtenidos con bases de datos de secuencias virales existentes. Esto permite realizar la asignación taxonómica, es decir, identificar a qué virus pertenecen las secuencias. Se utilizan herramientas bioinformáticas (Tabla 3) especializadas que buscan similitudes y coincidencias entre los contigs y las secuencias virales conocidas en las bases de datos, entre otras. 166,167.
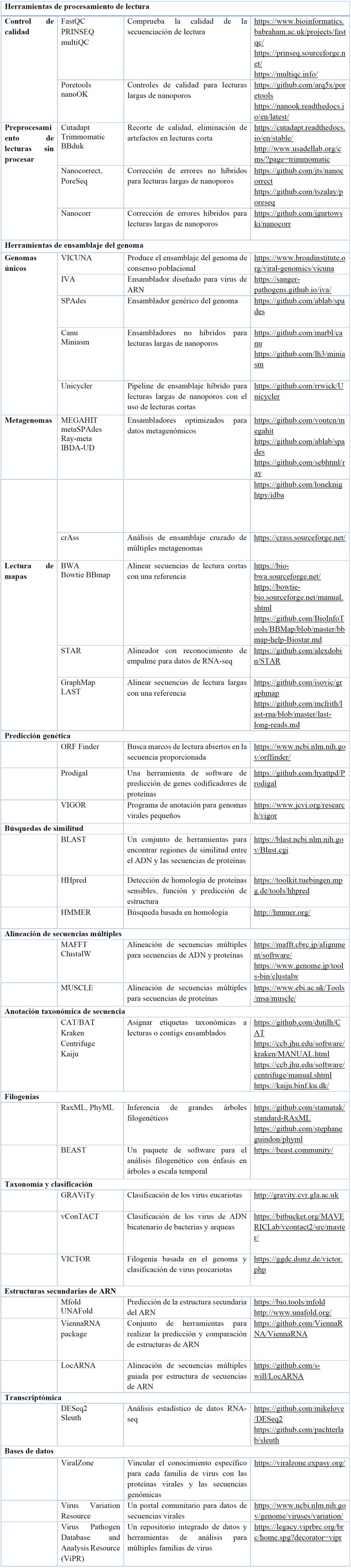
Tabla 3. Lista de herramientas y recursos de software seleccionados para tareas de bioinformática de virus
CONCLUSIONES
El tabaco (N. tabacum) es una de las especies vegetales con mayor susceptibilidad a patógenos, encontrándose más de 60 reportes de al menos 20 géneros virales. Dentro de este grupo existen virus de importancia económica por los daños que causan al hospedante, entre los que destacan TMV(Tobamovirus), CMV (Cucumovirus), PVY, TVMV y TEV (todos tres Potyvirus), TLCV (Begomovirus), y TSWV (Tospovirus). La mayoría de los virus que afectan plantas de tabaco principalmente son transmitidos por insectos, pero también por hongos, nematodos, semillas, polen y de forma mecánica. La incidencia de estos patógenos virales parece depender de diferentes factores como del genotipo usado, la edad de las plantas, prácticas de manejo, entre otros componentes, alcanzando valores entre 0,8 y 100%. Los fitovirus son identificados o caracterizados a través de pruebas de transmisión, técnicas bioquímicas y de microscopía electrónica, así como las relacionadas a las proteínas de la cápside viral como las técnicas de ELISA, microscopía inmunoelectrónica, inmunotransferencia, e inmunosensores de microbalanza de cristal de cuarzo. También, se los puede identificar usando diferentes variantes de PCR (nPCR, IC-PCR, M-PCR, PCRrt, RT-PCR, IC-RT-PCR y AmpliDet RNA), amplificación isotérmica (HAD, RPA, NASBA, RCA, LAMP y RT-LAMP), sistema de inmunidad procariótico de repeticiones palindrómicas cortas agrupadas regularmente interespaciadas (CRISPR), y secuenciación de nueva generación.
Conflictos de interés:
Los autores declaran no tener conflictos de intereses.
Contribución de autores
Todos los autores contribuyeron en la lectura, conceptualización, revisión y edición del manuscrito.
Financiación
Universidad Técnica de Manabí: BECA PARA EL DESARROLLO DE LA INVESTIGACIÓN A TRAVÉS DE SU TITULACIÓN.
REFERENCIAS
1. Russell, E. A. & Rahman, E. The Master Plant: Tobacco in Lowland South America. The Master Plant: Tobacco in Lowland South America (Bloomsbury Academic, 2015). doi:10.5040/9781474220279.
2. Chiurciu, I. A., Zaharia, I. & Soare, E. Aspects of the Tobacco Market Within the European Union and a Brief Moral Reflection on Smoking. Scientific Papers-Series Management Economic Engineering in Agriculture and Rural Development 19, 59–68 (2019).
3. Warner, K. E. The economics of Tobacco: myths and realities. Tob Control 9, 78–89 (2000). Doi: 10.1136/tc.9.1.78.
4. Akinyemi, I. A., Wang, F., Zhou, B., Qi, S. & Wu, Q. Ecogenomic survey of plant viruses infecting Tobacco by Next generation sequencing. Virol J 13, 181 (2016). Doi: 10.1186/s12985-016-0639-7.
5. Paximadis, M., Idris, A. M., Villarreal, A. & Rey, M. E. C. Characterization of tobacco Geminiviruses in the old and new world. 703–717 (1999).
6. Duke, M. Ethnicity, Well-Being, and the Organization of Labor among Shade Tobacco Workers. Med Anthropol 30, 409–424 (2011). Doi: 10.1080/01459740.2011.576727
7. Kamran, A. et al. Full genome sequence analysis and putative host-shifting of Milk vetch dwarf virus infecting Tobacco (Nicotiana tabacum) in China. Virol J 16, 1–5 (2019). Doi: 10.1186/s12985-019-1129-5
8. Takakura, Y., Udagawa, H., Shinjo, A. & Koga, K. Mutation of a Nicotiana tabacum L. eukaryotic translation-initiation factor gene reduces susceptibility to a resistance-breaking strain of Potato virus Y. Mol Plant Pathol 19, 2124–2133 (2018). Doi: 10.1111/mpp.12686.
9. Wang, S., Cui, W., Wu, X., Yuan, Q., Zhao, J., Zheng, H., … & Yan, F. Suppression of nbe-miR166h-p5 attenuates leaf yellowing symptoms of potato virus X on Nicotiana benthamiana and reduces virus accumulation. Mol Plant Pathol 19, 2384–2396 (2018). Doi: 10.1111/mpp.12717.
10. Zellner, W., Frantz, J. & Leisner, S. Silicon delays Tobacco ringspot virus systemic symptoms in Nicotiana tabacum. J Plant Physiol 168, 1866–1869 (2011). Doi: 10.1016/j.jplph.2011.04.002.
11. Cheng, L., Chen, X., Jiang, C., Ma, B., Ren, M., Cheng, Y., Liu, D., Geng, R. & Yang, A. High-density SNP genetic linkage map construction and quantitative trait locus mapping for resistance to cucumber mosaic virus in Tobacco (Nicotiana tabacum L.). Crop Journal 7, 539–547 (2019). Doi: 10.1016/j.cj.2018.11.010.
12. Dhawi, F. El-Beltagi, H. S., Abdel-Mobdy, Y, Salah, S., Ghaly, I. S., Abdel-Rahim, E. A., Mohamed, H. I. & Soliman, A. M. Synergistic impact of the pomegranate peels and its nanoparticles against the infection of tobacco mosaic virus (TMV). Fresenius Environ Bull 30, 731–746 (2021).
13. Gao, R., Tian, Y. P., Wang, J., Yin, X., Li, X. D., & Valkonen, J. P. Construction of an infectious cDNA clone and gene expression vector of Tobacco vein banding mosaic virus (genus Potyvirus). VirusRes 169, 276–281 (2012). Doi: 10.1016/j.virusres.2012.07.010.
14. Konakalla, N. C., Bag, S., Deraniyagala, A. S., Culbreath, A. K. & Pappu, H. R. Induction of plant resistance in Tobacco (Nicotiana tabacum) against Tomato spotted wilt orthotospovirus through foliar application of dsRNA. Viruses 13, (2021). Doi. 10.3390/v13040662.
15. Hidayat, S. H., Chatchawankanpanich, O. & Aidawati, N. Molecular Identification and Sequence Analysis of Tobacco Leaf Curl Begomovirus from Jember, East Java, Indonesia. Hayati 15, 13–17 (2008). Doi: 10.4308/hjb.15.1.13
16. Haňcinský, R., Mihálik, D., Mrkvová, M., Candresse, T. & Glasa, M. Plant viruses infecting solanaceae family members in the cultivated and wild environments: A review. Plants 9, (2020). Doi: 10.4308/hjb.15.1.13.
17. Gao, F. Liu, X., Du, Z., Hou, H., Wang, X., Wang, F., & Yang, J. Bayesian phylodynamic analysis reveals the dispersal patterns of tobacco mosaic virus in China. Virology 528, 110–117 (2019). Doi: 10.1016/j.virol.2018.12.001
18. Dai, J., Peng, H., Chen, W., Cheng, J. & Wu, Y. Development of multiplex real-time PCR for simultaneous detection of three Potyviruses in tobacco plants. J Appl Microbiol 114, 502–508 (2012). Doi: 10.1111/jam.12071.
19. Zanardo, L. G., de Souza, G. B. & Alves, M. S. Transcriptomics of plant–virus interactions: a review. Theor Exp Plant Physiol 31, 103–125 (2019). Doi: 10.1007/s40626-019-00143-z.
20. Zwart, M. P. & Elena, S. F. Modeling multipartite virus evolution: The genome formula facilitates rapid adaptation to heterogeneous environments. Virus Evol 6, 1–13 (2020). Doi: 10.1093/VE/VEAA022.
21. Ormeño, J., Sepúlveda, P., Rojas, R. & Araya, J. E. Datura Genus Weeds as an Epidemiological Factor of Alfalfa mosaic virus (AMV), Cucumber mosaic virus (CMV), and Potato virus Y (PVY) on Solanaceus Crops. Agricultura Técnica 66, 333–341 (2006). Doi: 10.4067/S0365-28072006000400001.
22. Moury, B. & Verdin, E. Viruses of Pepper Crops in the Mediterranean Basin. in Advances in Virus Research vol. 84 127–162 (2012). Doi: 10.1016/B978-0-12-394314-9.00004-X.
23. Xu, H. & Nie, J. Identification, characterization, and molecular detection of Alfalfa mosaic virus in Potato. Phytopathology 96, 1237–1242 (2006). Doi: 10.1094/PHYTO-96-1237.
24. Gao, Z., Pu, H., Liu, J., Wang, X., Zhong, C., Yue, N., … & Zhang, Y. Tobacco necrosis virus-AC single coat protein amino acid substitutions determine host-specific systemic infections of Nicotiana benthamiana and soybean. Molecular Plant-Microbe Interactions 34, 49–61 (2021). Doi: 10.1094/MPMI-07-20-0184-R.
25. Varanda, C. M. R. et al. A degenerate pair of primers for simultaneous detection of four alpha- and betanecroviruses. J Virol Methods 208, 63–65 (2014). Doi: 10.1016/j.jviromet.2014.08.004.
26. Babaei, G., Massah, A. & Koohi Habibi, M. Efficient translation of Eggplant mottled dwarf nucleorhabdovirus N and X genes requires both 5′ and 3′ UTRs. Virol J 18, 1–9 (2021). Doi: 10.1186/s12985-021-01601-4.
27. Fauquet, C. M. et al. Geminivirus strain demarcation and nomenclature. Arch Virol 153, 783–821 (2008). Doi: 0.1007/s00705-008-0037-6.
28. Goth, R. W., Ellis, P. J., De Villiers, G., Goins, E. W. & Wright, N. S. Characteristics and distribution of Potato latent carlavirus (Red LaSoda virus) in North America. Plant Dis 83, 751–753 (1999). Doi: 10.1094/PDIS.1999.83.8.751.
29. Kumar Tiwari, R., Kumar, R., Kaundal, P., Sharma, S. & Chakrabarti, S. Potato viruses and their diagnostic techniques: An overview. J Pharmacogn Phytochem 8, 1932–1944 (2019).
30. Loebenstein, G., Berger, P. H., Brunt, A. A. & Lawson, R. H. Virus and Virus-like Diseases of Potatoes and Production of Seed-Potatoes. Springer Science & Business Media (Springer Netherlands, 2001). Doi:10.1007/978-94-007-0842-6.
31. Sastry, K. S., Mandal, B., Hammond, J., Scott, S. W. & Briddon, R. W. Allium ampeloprasum var. porrum (Leek). in Encyclopedia of Plant Viruses and Viroids 53–57 (Springer India, 2019). Doi:10.1007/978-81-322-3912-3_26.
32. Wang, F, Qi, S., Gao, Z., Akinyemi, I. A., Xu, D., & Zhou, B. Complete genome sequence of Tobacco virus 1, a Closterovirus from Nicotiana tabacum. Arch Virol 161, 1087–1090 (2016). Doi: 10.1007/s00705-015-2739-x.
33. Fiallo-Olivé, E., Martínez-Zubiau, Y., Hernández-Zepeda, C., Carrillo-Trip, J. & Rivera-Bustamante, R. Identification of New Begomoviruses in Cuba Using Rolling Circle Amplification. Rev Prot Veg 24, 81–86 (2009).
34. Wisler, G. C., Duffus, J. E., Liu, H. & Li, R. H. Ecology and Epidemiology of Whitefly – Transmitted Closteroviruses. Plant Dis 82, 270–280 (1998).
35. Tungadi, T., Groen, S. C., Murphy, A. M., Pate, A. E., Iqbal, J., Bruce, T. J., Cunniffe, N. J. & Carr, J. P. Cucumber mosaic virus and its 2b protein alter emission of host volatile organic compounds but not aphid vector settling in Tobacco. Virol J 14, 1–9 (2017). Doi: 10.1186/s12985-017-0754-0.
36. Tungadi, T., Donnelly, R., Qing, L., Iqbal, J., Murphy, A. M., Pate, A. E., Cunniffe, N. J. & Carr, J. P. Cucumber mosaic virus 2b proteins inhibit virus-induced aphid resistance in Tobacco. Mol Plant Pathol 21, 250–257 (2019). Doi: 10.1111/mpp.12892.
37. Kameya-Iwaki, M., Murakami, K., Ito, S., Hanada, K. & Tanaka, S. Semipersistency of Myzus persicae Transmission of Cucumoviruses Systemically Infecting Leguminous Plants. Journal of General Plant Pathology 66, 64–67 (2000). Doi: 10.1007/pl00012922.
38. Ferreiro, C., Ostrówka, K., López-Moya, J. J. & Díaz-Ruíz, J. R. Nucleotide sequence and symptom modulating analysis of a peanut stunt virus-associated satellite RNA from Poland: High level of sequence identity with the American PSV satellites. Eur J Plant Pathol 102, 779–786 (1996). Doi: 10.1016/j.jviromet.2021.114277.
39. Obrȩpalska-Stȩplowska, A., Budziszewska, M., Wieczorek, P. & Czerwoniec, A. Analysis of two strains of Peanut stunt virus: SatRNA-associated and satRNA free. Virus Genes 44, 513–521 (2012). Doi: 10.1007/s11262-012-0729-6.
40. Sharman, M., Thomas, J. E. & Persley, D. M. Natural host range, thrips and seed transmission of distinct Tobacco streak virus strains in Queensland, Australia. Annals of Applied Biology 167, 197–207 (2015). Doi: 10.1111/aab.12218.
41. Pradeep, K. K., Satya, V. K., Selvapriya, M., Vijayasamundeeswari, A., Ladhalakshmi, D., Paranidharan, V., Rabindran, R., Samiyappan, R, Balasubramanian, P. & Velazhahan, R. Engineering resistance against Tobacco streak virus (TSV) in sunflower and Tobacco using RNA interference. Biol Plant 56, 735–741 (2012). Doi: 10.1007/s10535-012-0111-5.
42. Khalid, S., Zia-ur-Rehman, M., Hameed, U., Saeed, F., Khan, F., & Haider, M. S. Transmission specificity and coinfection of Mastrevirus with Begomovirus. Int J Agric Biol 19, 105–113 (2017). Doi: 10.17957/IJAB/15.0249.
43. Ashraf, M. et al. Influence of entomopathogenic fungus, Metarhizium anisopliae, alone and in combination with diatomaceous earth and thiamethoxam on mortality, progeny production, mycosis, and sporulation of the stored grain insect pests. Environmental Science and Pollution Research 24, 28165–28174 (2017). Doi: 10.1007/s11356-017-0383-6.
44. Karapetsi, L., Chatzivassiliou, E. K., Katis, N. I. & Maliogka, V. I. Artichoke yellow ringspot virusas the causal agent of a new viral disease of lettuce: Epidemiology and molecular variability. Plant Pathol70, 594–603 (2021). Doi: 10.1111/ppa.13309.
45. Gallitelli, D., Mascia, T. & Martelli, G. P. Viruses in Artichoke. Advances in Virus Research vol. 84 (Elsevier Inc., 2012).
46. Zarzyńska-Nowak, A., Ferriol, I., Falk, B. W., Borodynko-Filas, N. & Hasiów-Jaroszewska, B. Construction of Agrobacterium tumefaciens-mediated Tomato black ring virus infectious cDNA clones. Virus Res 230, 59–62 (2017). Doi: 10.1016/j.virusres.2017.01.014.
47. Šneideris, D. & Staniulis, J. Phylogenetic analysis of Lithuanian tomato black ring virus isolates. Zemdirbyste-Agriculture 101, 193–198 (2014). Doi: 10.1016/j.jviromet.2021.114277.
48. Samuitienë, M., Zitikaitë, I., Navalinskienë, M. & Valiûnas, D. Identification of Tomato ringspot nepovirus by RT-PCR. 4, 35–38 (2021).
49. Renvoisé, J. P., Chambon, F., Gleize, M., Pradeilles, N., Garnier, S., & Rolland, M. Selection, optimization and characterization of molecular tests for the detection of Tobacco ringspot virus (TRSV). EPPO Bulletin 49, 111–121 (2019). Doi: 10.1111/epp.12554.
50. Rowhani, A., Daubert, S. D., Uyemoto, J. K., Al Rwahnih, M. & Fuchs, M. Grapevine viruses: Molecular biology, diagnostics and management. in Grapevine Viruses: Molecular Biology, Diagnostics and Management 109–126 (2017). Doi:10.1007/978-3-319-57706-7.
51. Wang, R. Y., Kritzman, A., Hershman, D. E. & Ghabrial, S. A. Aphis glycines as a vector of persistently and nonpersistently transmitted viruses and potential risks for soybean and other crops. Plant Dis 90, 920–926 (2006). Doi: 10.1094/PD-90-0920.
52. Huang, C. et al. Transcriptomic profile of Tobacco in response to Tomato zonate spot orthotospovirus infection. Virol J 14, 1–11 (2017). Doi: 10.1186/s12985-017-0821-6
53. Zheng, X., Zhang, J., Chen, Y., Dong, J. & Zhang, Z. Effects of tomato zonate spot virus infection on the development and reproduction of its vector Frankliniella occidentalis (Thysanoptera: Thripidae). Florida Entomologist 97, 549–554 (2014). Doi: 10.1896/054.097.0228.
54. Wu, X., Wu, X., Li, W. & Cheng, X. Molecular characterization of a divergent strain of Calla lily chlorotic spot virus infecting cetulce (Lactuca sativa var. augustana) in China. Arch Virol 163, 1375–1378 (2018). Doi: 10.1007/s00705-018-3743-8.
55. Kuo, Y. W., Gilbertson, R. L., Turini, T., Brennan, E. B., Smith, R. F., & Koike, S. T. Characterization and epidemiology of outbreaks of Impatiens necrotic spot virus on lettuce in Coastal California. Plant Dis 98, 1050–1059 (2014). Doi: 10.1094/PDIS-07-13-0681-RE.
56. Martínez-Ochoa, N., Csinos, A. S., Whitty, E. B., Johnson, A. W. & Parrish, M. J. First Report on the Incidence of Mixed Infections of Impatiens necrotic spot virus (INSV) and Tomato spotted wilt virus (TSWV) in Tobacco Grown in Georgia, South Carolina, and Virginia . Plant Health Prog 4, 40 (2003). Doi: 10.1094/php-2003-0417-01-hn.
57. Zarzyńska-Nowak, A., Hasiów-Jaroszewska, B., Korbecka-Glinka, G., Przybyś, M. & Borodynko-Filas, N. A multiplex RT-PCR assay for simultaneous detection of Tomato spotted wilt virus and Tomato yellow ring virus in tomato plants. Canadian Journal of Plant Pathology 40, 580–586 (2018). Doi: 10.1080/07060661.2018.1503195.
58. Tan, S. T., Liu, F., Lv, J., Liu, Q. L., Luo, H. M., Xu, Y., Ma, Y., Chen, X., Lan, P., Chen, H., Cao, M. & Li, F. et al. Identification of two novel poleroviruses and the occurrence of Tobacco bushy top disease causal agents in natural plants. Sci Rep 11, 1–14 (2021). Doi: 10.1038/s41598-021-99320-x.
59. Mo, X. H., Chen, Z. B. & Chen, J. P. Complete nucleotide sequence and genome organization of a Chinese isolate of Tobacco vein distorting virus. Virus Genes 41, 425–431 (2010). Doi: 10.1007/s11262-010-0524-1.
60. Mo, X. H., Chen, Z. B. & Chen, J. P. Molecular identification and phylogenetic analysis of a viral RNA associated with the Chinese tobacco bushy top disease complex. Annals of Applied Biology 158, 188–193 (2011). Doi: 10.1111/j.1744-7348.2010.00452.x.
61. Wang, Q., Mao, J. J., Xiang, H. Y., Dong, L. H., Sun, Y. H., Liu, G. S., & Liu, H. B. First report of Brassica yellows virus on Tobacco in China. Plant Dis 99, 1192 (2015). Doi: 10.1094/PDIS-04-15-0426-PDN.
62. Wang, Q. Xu, F. Z., An, L. L., Xiang, H. Y., Zhang, W. H., Liu, G. S., & Liu, H. B. Molecular characterization of a new recombinant Brassica yellows virus infecting Tobacco in China. Virus Genes 55, 253–256 (2019). Doi: 10.1007/s11262-019-01636-4.
63. Zuo, D. P., He, M. J., Chen, X. R., Hu, R. J., Zhao, T. Y., Zhang, X. Y., … & Han, C. G. A simple method for the acquisition and transmission of Brassica yellows virus from transgenic plants and frozen infected leaves by aphids. Plants 10, (2021). Doi: 10.3390/plants10091944.
64. Milošević, D., Ignjatov, M., Marjanović-Jeromela, A., Stanković, I., Nikolić, Z., Tamindžić, G., & Krstić, B. Molecular characterization of Turnip yellows virus– a new pathogen of mustard in Serbia. Ratar.Povrt. 56, 82–87 (2019). Doi: 10.5937/ratpov56-23514.
65. Wang, F., Wu, Q. F., Zhou, B. G., Gao, Z. L. & Xu, D. F. First Report of Turnip yellows virus in Tobacco in China. Plant Dis 99, 1870–1870 (2015). Doi: 10.1094/PDIS-04-15-0426-PDN
66. Alcaide, C., Rabadán, M. P., Juárez, M. & Gómez, P. Long-term cocirculation of two strains of Pepino mosaic virus in tomato crops and its effect on population genetic variability. Phytopathology 110, 49–57 (2020). Doi: 10.1094/PHYTO-07-19-0247-FI.
67. Blystad, D. R., van der Vlugt, R., Alfaro-Fernández, A., del Carmen Córdoba, M., Bese, G., Hristova, D., Pospieszny, H., Mehle, N., Ravnikar, M., Tomassoli, L., Varveri, C. & Nielsen, S. L. Host range and symptomatology of Pepino mosaic virus strains occurring in Europe. Eur J Plant Pathol 143, 43–56 (2015). Doi: 10.1007/s10658-015-0664-1.
68. Shipp, J. L., Buitenhuis, R., Stobbs, L., Wang, K., Kim, W. S., & Ferguson, G. Vectoring of Pepino mosaic virus by bumble-bees in tomato greenhouses. Annals of Applied Biology 153, 149–155 (2008). Doi: 10.1111/j.1744-7348.2008.00245.x.
69. Noël, P., Hance, T. & Bragard, C. Transmission of the Pepino mosaic virus by whitefly. Eur J Plant Pathol 138, 23–27 (2014). Doi: 10.1007/s10658-013-0313-5.
70. Khan, A., Tabassum, B., Nasir, I. A., Bilal, M., Tariq, M., & Husnain, T. Potato virus X from Pakistan: Coat protein sequence analysis. J Anim Plant Sci 25, 1016–1021 (2015).
71. Verchot, J. Potato virus X : A global potato‐infecting virus and type member of the Potexvirus genus. Mol Plant Pathol 23, 315–320 (2022). Doi: 10.1111/mpp.13163.
72. Röder, J., Dickmeis, C. & Commandeur, U. Small, smaller, nano: New applications for potato virus X in nanotechnology. Front Plant Sci 10, 158 (2019). Doi: 10.3389/fpls.2019.00158
73. Rao, S., Chen, X., Qiu, S., Peng, J., Zheng, H., Lu, Y., Wu, G., Chen, J., Jiang, W., Zhang, Y. & Yan, F. Identification of Two New Isolates of Chilli veinal mottle virus from different regions in China: Molecular diversity, Phylogenetic and Recombination Analysis. Front Microbiol 11, 1–7 (2020). Doi: 10.3389/fmicb.2020.616171.
74. Ryšlavá, H., Müller, K., Semorádová, Š., Synková, H. & Čeřovská, N. Photosynthesis and activity of phosphoenolpyruvate carboxylase in Nicotiana tabacum L. leaves infected by Potato virus A and Potato virus Y. Photosynthetica 41, 357–363 (2003). Doi: 10.1111/pce.12752.
75. Ahmadvand, R., Takács, A., Taller, J., Wolf, I. & Polgár, Z. Potato viruses and resistance genes in potato. Acta Agronomica Hungarica 60, 283–298 (2012). Doi: 10.1556/AAgr.60.2012.3.10.
76. Fox, A., Collins, L. E., Macarthur, R., Blackburn, L. F. & Northing, P. New aphid vectors and efficiency of transmission of Potato virus A and strains of Potato virus Y in the UK. Plant Pathol 66, 325–335 (2017). Doi: 10.1111/ppa.12561.
77. Matsumoto, K., Yasaka, R., Setoyama, T., Kawano, S. & Ohshima, K. Chilli pepper rugose mosaic disease caused by Pepper veinal mottle virus occurs on Ishigaki Island, Japan. Journal of General Plant Pathology 82, 57–60 (2016). Doi: 10.1007/s10327-015-0634-7.
78. Moury, B., Palloix, A., Caranta, C., Gognalons, P., Souche, S., Selassie, K. G. Serological, molecular, and pathotype diversity of Pepper veinal mottle virus and Chili veinal mottle virus. Phytopathology 95, 227–232 (2005). Doi: 10.1094/PHYTO-95-0227.
79. Alegbejo, M. D. & Abo, M. E. Ecology, Epidemiology and Control of Pepper Veinal mottle virus (PVMV), Genus Potyvirus, in West Africa. Journal of Sustainable Agriculture 20, 5–16 (2002). Doi: 10.1300/J064v20n02_03.
80. Fajinmi, A. A. Pepper Veinal Mottle Virus, a Potyvirus of pepper under tropical conditions. International Journal of Vegetable Science 19, 150–156 (2013). Doi: 10.1080/19315260.2012.687439.
81. Mondal, S., Ghanim, M., Roberts, A. & Gray, S. M. Different potato virus Y strains frequently co-localize in single epidermal leaf cells and in the aphid stylet. Journal of General Virology 102, 001576 (2021). Doi: 10.1099/jgv.0.001576.
82. Faurez, F., Baldwin, T., Tribodet, M. & Jacquot, E. Identification of new Potato virus Y (PVY) molecular determinants for the induction of vein necrosis in Tobacco. Mol Plant Pathol 13, 948–959 (2012). Doi: 10.1111/j.1364-3703.2012.00803.x.
83. Ren, G., Wang, X. F., Chen, D., Wang, X. W., Fan, X. J., & Liu, X. D. Potato virus Y-infected Tobacco affects the growth, reproduction, and feeding behavior of a vector aphid, Myzus persicae(Hemiptera: Aphididae). Appl Entomol Zool 50, 239–243 (2015). Doi: 10.1007/s13355-015-0328-9.
84. Kirchner, S. M., Döring, T. F., Hiltunen, L. H., Virtanen, E. & Valkonen, J. P. T. Information-theory-based model selection for determining the main vector and period of transmission of Potato virusY. Annals of Applied Biology 159, 414–427 (2011). Doi: 10.1111/j.1744-7348.2011.00501.x.
85. Pelletier, Y., Nie, X., Giguère, M. A., Nanayakkara, U., Maw, E., & Foottit, R. A new approach for the identification of aphid vectors (Hemiptera: Aphididae) of Potato virus Y. J Econ Entomol 105, 1909–1914 (2012). Doi: 10.1603/EC12085.
86. Agudelo-Romero, P., de la Iglesia, F. & Elena, S. F. The pleiotropic cost of host-specialization in Tobacco etch potyvirus. Infection, Genetics and Evolution 8, 806–814 (2008). Doi: 10.1016/j.meegid.2008.07.010.
87. Ruiz-Ferrer, V., Boskovic, J., Alfonso, C., Rivas, G., Llorca, O., López-Abella, D., & López-Moya, J. J. Structural analysis of Tobacco etch potyvirus HC-Pro oligomers involved in aphid transmission. J Virol 79, 3758–3765 (2005). Doi: 10.1128/JVI.79.6.3758-3765.2005.
88. Lai, R., You, M., Lotz, L. A. P. (Bert) & Vasseur, L. Response of green peach aphids and other arthropods to garlic intercropped with Tobacco. Agron J 103, 856–863 (2011). Doi: 10.2134/agronj2010.0404.
89. Wei, S., He, X., Wang, D., Xiang, J., Yang, Y., Yuan, S., Shang, J. & Yang, H.. Genetic structure and variability of tobacco vein banding mosaic virus populations. Arch Virol 164, 2459–2467 (2019). Doi: 10.1007/s00705-019-04342-6.
90. Zhang, C. L., Gao, R., Wang, J., Zhang, G. M., Li, X. D., & Liu, H. T. Molecular variability of Tobacco vein banding mosaic virus populations. Virus Res 158, 188–198 (2011). Doi: 10.1016/j.virusres.2011.03.031.
91. Yu, X.-Q., Lan, Y. F., Wang, H. Y., Liu, J. L., Zhu, X. P., Valkonen, J. P., & Li, X. D. The complete genomic sequence of Tobacco vein banding mosaic virus and its similarities with other Potyviruses. Virus Genes 35, 801–806 (2007). Doi: 10.1007/s11262-007-0135-7.
92. Gadhave, K. R., Saurabh, G., Rasmussen, D. A. & Srinivasan, R. Aphid transmission of Potyvirus : The largest plant-infecting RNA virus. Viruses 12, 773 (2020). Doi: 10.3390/v12070773.
93. Salvador, B., Saenz, P., Yangüez, E., Quiot, J. B., Quiot, L., Delgadillo, M. O., García, J. & SIMÓN‐MATEO, C. Host-specific effect of P1 exchange between two Potyviruses. Mol Plant Pathol 9, 147–155 (2008). Doi: 10.1111/j.1364-3703.2007.00450.x.
94. Blawid, R., Rodrigues, K. B., de Moraes Rêgo, C., Inoue-Nagata, A. K. & Nagata, T. Complete genome sequence of Tobacco mosqueado virus. Arch Virol 161, 2619–2622 (2016). Doi: 10.1007/s00705-016-2956-y.
95. Nasim, N. & Dey, N. Pararetroviruses: Plant Infecting dsDNA Viruses. Plant Mol Biol Report 40, 106–118 (2022). Doi: 10.1007/s11105-021-01294-7.
96. Zhao, W., Wu, S., Du, L., Li, T., Cheng, Z., Zhou, Y., & Ji, Y. Development of a reverse-transcription loop-mediated isothermal amplification assay for the detection of Tobacco mild green mosaic virus (TMGMV). J Virol Methods 298, 114277 (2021). Doi: 10.1016/j.jviromet.2021.114277.
97. El-Gendi, H., Al-Askar, A. A., Király, L., Samy, M. A., Moawad, H., & Abdelkhalek, A. Foliar applications of Bacillus subtilis HA1 culture filtrate enhance tomato growth and induce systemic resistance against Tobacco mosaic virus Infection. Horticulturae 8, 1–21 (2022). Doi: 10.3390/horticulturae8040301.
98. Abo-Zaid, G. A., Matar, S. M. & Abdelkhalek, A. Induction of plant resistance against Tobacco mosaic virus using the biocontrol agent Streptomyces cellulosae isolate Actino 48. Agronomy 10, 1620 (2020). Doi: 10.3390/agronomy10111620.
99. Ellis, M. D., Hoak, J. M., Ellis, B. W., Brown, J. A., Sit, T. L., Wilkinson, C. A., Reed, T. D. & Welbaum, G. E. Quantitative real-time PCR analysis of individual flue-cured tobacco seeds and seedlings reveals seed transmission of Tobacco mosaic virus. Phytopathology 110, 194–205 (2020). Doi: 10.1094/PHYTO-06-19-0201-FI.
100. Ladipo, J. L., Koenig, R. & Lesemann, D. E. Nigerian tobacco latent virus: A new Tobamovirus from Tobacco in Nigeria. Eur J Plant Pathol 109, 373–379 (2003). Doi: 10.1023/A:1023557723943.
101. Mojtahedi, H., Santo, G. S., Thomas, P. E., Crosslin, J. M. & Boydston, R. A. Eliminating Tobacco rattle virus from viruliferous Paratrichodorus allius and Establishing a New Virus-Vector Combination. J Nematol 34, 66–9 (2002).
102. Otulak, K., Chouda, M., Chrzanowska, M. & Garbaczewska, G. Ultrastructural effects of infection caused by Tobacco rattle virus transmitted by Trichodorus primitivus in potato and tobacco tissues. Canadian Journal of Plant Pathology 34, 126–138 (2012). Doi: 10.1080/07060661.2012.665387.
103. Otulak, K., Chouda, M., Bujarski, J. & Garbaczewska, G. The evidence of Tobacco rattle virusimpact on host plant organelles ultrastructure. Micron 70, 7–20 (2015). Doi: 10.1016/j.micron.2014.11.007.
104. Elmhirst, J. F. & Forge, T. A. Winter cover crops as reservoirs of Tobacco rattle virus (TRV) and hosts of root-lesion, root knot and stubby-root nematodes. Canadian Journal of Plant Pathology 43, 631–640 (2021). Doi: 10.1080/07060661.2020.1867898.
105. Ribeiro, S., Kitajima, E., Oliveira, C. & Koenig, R. A Strain of Eggplant mosaic virus isolated from naturally infected tobacco plants in Brazil. Plant Disease (USA) vol. 80 446–449 Preprint at (1996).
106. Megias, E. et al. Chloroplast proteome of Nicotiana benthamiana infected by Tomato blistering mosaic virus. Protein Journal 37, 290–299 (2018). Doi: 10.1007/s10930-018-9775-9.
107. Syller, J. Molecular and biological features of Umbraviruses, the unusual plant viruses lacking genetic information for a capsid protein. Physiol Mol Plant Pathol 63, 35–46 (2003). Doi: 10.1016/j.pmpp.2003.08.004.
108. Zhang, J., Liu, X., Li, W., Zhang, J., Xiao, Z., Zhou, Z., Liu, T., Liu, Y., Wang, F., Zhang, S. & Yang, J. Rapid detection of Milk vetch dwarf virus by loop-mediated isothermal amplification. J Virol Methods 261, 147–152 (2018). Doi: 10.1016/j.jviromet.2018.08.012.
109. Yang, J. G., Wang, S. P., Liu, W., Li, Y., Shen, L. L., Qian, Y. M., Wang, F. L. & Du, Z. G. First report of Milk vetch dwarf virus associated with a disease of Nicotiana tabacum in China. Plant Diseasevol. 100 Preprint at https://doi.org/10.1094/PDIS-11-15-1295-PDN (2016). Doi: 10.1094/PDIS-11-15-1295-PDN.
110. Applied Plant Virology: Advances, Detection, and Antiviral Strategies. (2020).
111. Ding, X. S., Liu, J., Cheng, N. H., Folimonov, A., Hou, Y. M., Bao, Y., Katagi, C., Carter, S. A. & Nelson, R. S. The Tobacco mosaic virus 126-kDa Protein Associated with Virus Replication and Movement Suppresses RNA Silencing. / 583 MPMI vol. 17 (2004). Doi: 10.1094/MPMI.2004.17.6.583.
112. Höller, K., Király, L., Künstler, A., Müller, M., Gullner, G., Fattinger, M., & Zechmann, B. Enhanced glutathione metabolism is correlated with sulfur-induced resistance in Tobacco mosaic virus-infected genetically susceptible Nicotiana tabacum Plants. Mol Plant Microbe Interact 23, 1448–1459 (2010). Doi: 10.1094/MPMI.
113. Das, P. P., Lin, Q. & Wong, S. M. Comparative proteomics of Tobacco mosaic virus-infected Nicotiana tabacum plants identified major host proteins involved in photosystems and plant defence. J Proteomics 194, 191–199 (2019). Doi: 10.1016/j.jprot.2018.11.018.
114. Wilhelmová, N., Procházková, D., Šindelářová, M. & Šindelář, L. Photosynthesis in leaves of Nicotiana tabacum L. infected with Tobacco mosaic virus. PHOTOSYNTHETICA vol. 43 (2005).
115. Liu, D., Zhao, Q., Cheng, Y., Li, D., Jiang, C., Cheng, L., Li, D., Jiang, C., Cheng, L., Wang, Y. & Yang, A. Transcriptome analysis of two cultivars of Tobacco in response to Cucumber mosaic virusinfection. Sci Rep 9, (2019). Doi: 10.1038/s41598-019-39734-w.
116. Lu, J., Du, Z. X., Kong, J., Chen, L. N., Qiu, Y. H., Li, G. F., Meng, X. H. & Zhu, S. F.Transcriptome Analysis of Nicotiana tabacum infected by Cucumber mosaic virus during systemic symptom development. PLoS One 7, e43447 (2012). Doi: 10.1371/journal.pone.0043447.
117. Mochizuki, T. & Ohki, S. T. Cucumber mosaic virus: Viral genes as virulence determinants. Mol Plant Pathol 13, 217–225 (2012). Doi: 10.1111/j.1364-3703.2011.00749.x.
118. Velasquez, N., Hossain, M. J. & Murphy, J. F. Differential disease symptoms and full-length genome sequence analysis for three strains of Tobacco etch virus. Virus Genes 50, 442–449 (2015). Doi: 10.1007/s11262-014-1146-9.
119. Tribodet, M., Glais, L., Kerlan, C. & Jacquot, E. Characterization of Potato virus Y (PVY) molecular determinants involved in the vein necrosis symptom induced by PVYN isolates in infected Nicotiana tabacum cv. Xanthi. Journal of General Virology 86, 2101–2105 (2005). Doi. 10.1099/vir.0.80926-0.
120. Chen, S., Li, F., Liu, D., Jiang, C., Cui, L., Shen, L., Liu, G. & Yang, A. Dynamic expression analysis of early response genes induced by Potato virus Y in PVY-resistant Nicotiana tabacum. Plant Cell Rep 36, 297–311 (2017). Doi: 10.1007/s00299-016-2080-1.
121. Synková, H., Semorádová, Š., Schnablová, R., Müller, K., Pospíšilová, J., Ryšlavá, H., Malbeck, J. & Čeřovská, N. Effects of biotic stress caused by Potato virus Y on photosynthesis in ipt transgenic and control Nicotiana tabacum L. Plant Science 171, 607–616 (2006). Doi: 10.1016/j.plantsci.2006.06.002.
122. Mayunga, D. S. & Kapooria, R. G. Incidence and identification of virus diseases of Tobacco in three provinces of Zambia. (2003). Bulletin OEPP/EPPO Bulletin. Report
123. Gonzalez-Jara, P., Tenllado, F., Martínez‐García, B., Atencio, F. A., Barajas, D., Vargas, M., Diaz-Ruiz, J. & Díaz‐Ruíz, J. R. Host-dependent differences during synergistic infection by Potyviruses with Potato virus X. Mol Plant Pathol 5, 29–35 (2004). Doi: 10.1111/j.1364-3703.2004.00202.x.
124. Senanayake, D. M. J. B. & Mandal, B. Expression of symptoms, viral coat protein and silencing suppressor gene during mixed infection of a N–Wi strain of Potato virus Y and an asymptomatic strain of Potato virus X. Virusdisease 25, 314–321 (2014). Doi: 10.1007/s13337-014-0204-1.
125. Sheng, Y., Yang, L., Li, C., Wang, Y. & Guo, H. Transcriptomic changes in Nicotiana tabacumleaves during mosaic virus infection. 3 Biotech 9, 1–13 (2019). Doi: 10.1007/s13205-019-1740-6.
126. Sarwar, M. Mite (Acari Acarina) vectors involved in transmission of plant viruses. Applied Plant Virology (INC, 2020). Doi:10.1016/b978-0-12-818654-1.00020-7.
127. Herrbach, E. & Chesnais, Q. Vector transmission of plant viruses. Encyclopedia of Virology vol. 3 (2021). Doi: 10.1016/b978-0-12-809633-8.21349-6.
128. Shi, X., Zhang, Z., Zhang, C., Zhou, X., Zhang, D., & Liu, Y. The molecular mechanism of efficient transmission of plant viruses in variable virus–vector–plant interactions. Hortic Plant J 7, 501–508 (2021). Doi: 10.1016/j.hpj.2021.04.006.
129. Singh, S., Awasthi, L. P., Jangre, A. & Nirmalkar, V. K. Transmission of plant viruses through soil-inhabiting nematode vectors. Applied Plant Virology (INC, 2020). Doi:10.1016/b978-0-12-818654-1.00022-0.
130. Nie, X., Singh, M., Chen, D., Gilchrist, C., Soqrat, Y., Shukla, M., Creelman, A., Dickison, V., Nie, B., Lavoie, J. & Bisht, V. Development of High-Resolution DNA melting analysis for simultaneous detection of Potato mop-top virus and its vector Spongospora subterranea, in Soil. Plant Dis 105, 948–957 (2021). Doi: 10.1094/PDIS-06-20-1321-RE.
131. Turan, V., Verma, S., Sharma, P., Katna, S. & Dev, B. Major vectors of the plant viruses: a review. J Entomol Zool Stud 8, 1365–1370 (2020).
132. Whitfield, A. E., Falk, B. W. & Rotenberg, D. Insect vector-mediated transmission of plant viruses. Virology 479–480, 278–289 (2015). Doi: 10.1016/j.virol.2015.03.026.
133. Ziegler-Graff, V. Molecular insights into host and vector manipulation by plant viruses. Viruses 12, (2020). Doi: 10.3390/v12030263.
134. Pinheiro-Lima, B., Pereira-Carvalho, R. C., Alves-Freitas, D. M., Kitajima, E. W., Vidal, A. H., Lacorte, C., Godinho, M. T., Fontenele, R. S., Faria, J. C., Abreu, E. F. M., Varsani, A., Ribeiro, S. G. & Melo, F. L. Transmission of the Bean-Associated Cytorhabdovirus by the Whitefly Bemisia tabaciMEAM1. Viruses 12, (2020). Doi: 10.3390/v12091028.
135. Fereres, A. & Raccah, B. Plant virus transmission by insects. eLS 1–12 (2015) Doi: 10.1002/9780470015902.a0000760.pub3.
136. Fiallo-Olivé, E. & Navas-Castillo, J. Tomato chlorosis virus, an emergent plant virus still expanding its geographical and host ranges. Mol Plant Pathol 20, 1307–1320 (2019). Doi: 10.1111/mpp.12847.
137. Asadhi, S., Padmavati, S., Visvavidyalayam, M., Yeturu, S. & Usha, R. Partial Characterization of Replicase Gene of Tobacco streak ilarvirus in Onion (Allium cepa.L ). Article in International Journal of Applied Biology and Pharmaceutical Technology (2015).
138. Gaafar, Y. Z. A. et al. Caraway yellows virus, a novel Nepovirus from Carum carvi. Virol J 16, 4–9 (2019). Doi: 10.1186/s12985-019-1181.
139. Dietzgen, R. G., Mann, K. S. & Johnson, K. N. Plant virus-insect vector interactions: Current and potential future research directions. Viruses 8, 1–21 (2016). Doi: 10.3390/v8110303.
140. Güneş, N., Türkseven, S. G., Özsari, pınar, Gümüş, mustafa & Baysal Sivritepe, D. Incidence and possible sources of Tomato spotted wilt virus in Tobacco grown in Denizli Province, Turkey. Not Bot Horti Agrobot Cluj Napoca 50, 12529 (2022). Doi: 10.15835/nbha50212529.
141. Gunay, A. & Usta, M. First investigation of five Tobacco viruses using PCR based methods in tobacco plants grown in Adiyaman, Turkey. (2020). Report.
142. Ding, M., Yang, C., Zhang, L., Jiang, Z. L., Fang, Q., Qin, X. Y., & Zhang, Z. K. Occurrence of Chilli veinal mottle virus in Nicotiana tabacum in Yunnan, China. https://doi.org/10.1094/PDIS-09-10-0686 95, 357 (2011).
143. Khateri, H., Moarrefzadeh, N., Koohi-Habibi, M., Mosahebi, G., Hosseini, A., & Hamzeh, N. High incidence of Tobacco streak virus in the tobacco fields of Iran. Commun Agric Appl Biol Sci 71, 1213–1216 (2006).
144. Delić, D., D., Balech, B., Radulović, M., Đurić, Z., Lolić, B., Santamaria, M., & Đurić, G. Molecular identification of Tomato spotted wilt virus on pepper and Tobacco in Republic of Srpska (Bosnia and Herzegovina). Eur J Plant Pathol 150, 785–789 (2018). Doi: 10.1007/s10658-017-1313-7.
145. Chatzivassiliou, E. K., Efthimiou, K., Drossos, E., Papadopoulou, A., Poimenidis, G., & Katis, N. I. A survey of tobacco viruses in tobacco crops and native flora in Greece. Eur J Plant Pathol 110, 1011–1023 (2004). Doi: 10.1007/s10658-004-1598-1.
146. Kumlachew, A. Detection of diseases, identification and diversity of viruses: A Review. J Biol Agric Healthc 5, 204–214 (2015).
147. Rubio, L., Galipienso, L. & Ferriol, I. Detection of plant viruses and disease management: relevance of genetic diversity and evolution. Front Plant Sci 11, 1092 (2020). Doi: 10.3389/fpls.2020.01092.
148. Fang, Y. & Ramasamy, R. P. Current and prospective methods for plant disease detection. Biosensors (Basel) 5, 537–561 (2015). Doi: 0.3390/bios5030537.
149. Mehetre, G. T. et al. Current developments and challenges in plant viral diagnostics: A systematic review. Viruses 13, 1–31 (2021). Doi: 0.3390/bios5030537.
150. Bhat, A. I. & Rao, G. P. Polymerase Chain Reaction. in Characterization of Plant Viruses, Springer Protocols Handbooks 323–346 (2020). Doi:10.1007/978-1-0716-0334-5_7.
151. Serçe, Ç. U. & Ayyaz, M. Diagnosis of the casual viruses of crop plants. in Applied Plant Virology: Advances, Detection, and Antiviral Strategies 55–67 (2020). Doi:10.1016/B978-0-12-818654-1.00004-9.
152. Kumar, R., Kaundal, P., Tiwari, R. K., Lal, M. K., Kumari, H., Kumar, R., Chandra Naga, K., Kumar, A., Singh, B.,Sagar, V. & Sharma, S. Development of Reverse Transcription Recombinase Polymerase Amplification (RT-RPA): A methodology for quick diagnosis of Potato leafroll viral disease in potato. International Journal of Molecular Sciences 2023, Vol. 24, Page 2511 24, 2511 (2023). Doi: 10.3390/ijms24032511.
153. Pallás, V., Sánchez-Navarro, J. A. & James, D. Recent advances on the multiplex molecular detection of plant viruses and viroids. Front Microbiol 9, 1–11 (2018). Doi: 10.3389/fmicb.2018.02087.
154. Letschert, B., Adam, G., Lesemann, D. E., Willingmann, P. & Heinze, C. Detection and differentiation of serologically cross-reacting Tobamoviruses of economical importance by RT-PCR and RT-PCR-RFLP. J Virol Methods 106, 1–10 (2002). Doi: 10.1016/S0166-0934(02)00135-0.
155. Liu, F., Tan, G., Li, X., Chen, H., Li, R., & Li, F. Simultaneous detection of four causal agents of Tobacco bushy top disease by a multiplex one-step RT-PCR. J Virol Methods 205, 99–103 (2014). Doi: 10.1016/j.jviromet.2014.05.003.
156. Chalam, V. C., Sharma, V. D., Sharma, R. & Maurya, A. K. Modern technologies for the diagnosis and assay of plants viruses. in Applied Plant Virology: Advances, Detection, and Antiviral Strategies 69–77 (INC, 2020). Doi:10.1016/B978-0-12-818654-1.00005-0.
157. Lee, H. J., Cho, I. S., Ju, H. J. & Jeong, R. D. Rapid and visual detection of Tomato spotted wilt virus using recombinase polymerase amplification combined with lateral flow strips. Mol Cell Probes 57, 1–4 (2021). Doi: 10.1016/j.mcp.2021.101727.
158. Rasni, W. H. N. W., Yahaya, N. & Mohamed Rehan, M. Recombinase polymerase amplification and their application in phytopathogen detection. Malaysian Journal of Science Health & Technology 8, 14–24 (2022). Doi: 10.33102/2022254.
159. Panno, S., Matić, S., Tiberini, A., Caruso, A. G., Bella, P., Torta, L., Stassi, R. & Davino, S. Loop mediated isothermal amplification: Principles and applications in plant virology. Plants 9, 1–28 (2020). Doi: 10.3390/plants9040461.
160. Soroka, M., Wasowicz, B. & Rymaszewska, A. Loop-mediated isothermal amplification (Lamp): The better sibling of PCR? Cells 10, (2021). Doi: 10.3390/cells10081931.
161. Budziszewska, M., Wieczorek, P. & Obrępalska-Stęplowska, A. One-step reverse transcription loop-mediated isothermal amplification (RT-LAMP) for detection of Tomato torrado virus. Arch Virol161, 1359–1364 (2016). Doi: 10.1007/s00705-016-2774-2.
162. Zhang, C., Liu, S., Li, X., Zhang, R. & Li, J. Virus-Induced Gene Editing and Its Applications in Plants. International Journal of Molecular Sciences vol. 23 Preprint at https://doi.org/10.3390/ijms231810202 (2022).
163. Kalinina, N. O., Khromov, A., Love, A. J. & Taliansky, M. E. CRISPR applications in plant virology: Virus resistance and beyond. Phytopathology 110, 18–28 (2020). Doi: 10.1094/PHYTO-07-19-0267-IA.
164. Mahas, A., Hassan, N., Aman, R., Marsic, T., Wang, Q., Ali, Z., & Mahfouz, M. M. Lamp-coupled CRISPR–CAS12a module for rapid and sensitive detection of plant DNA viruses. Viruses 13, (2021). Doi: 10.3390/v13030466.
165. Rott, M., Xiang, Y., Boyes, I., Belton, M., Saeed, H., Kesanakurti, P., Hayes, S., Lawrence, T., Birch, C., Bhagwat, B. & Rast, H. Application of next generation sequencing for diagnostic testing of tree fruit viruses and viroids. Plant Dis 101, 1489–1499 (2017). Doi: 10.1094/PDIS-03-17-0306-RE
166. Adams, I. & Fox, A. Diagnosis of Plant Viruses Using Next- Generation Sequencing and Metagenomic Analysis. in Current Research Topics in Plant Virology 323–335 (2016). Doi:10.1007/978-3-319-32919-2.
167. Barba, M., Czosnek, H. & Hadidi, A. Historical perspective, development and applications of next-generation sequencing in plant virology. Viruses 6, 106–136 (2014). Doi: 10.3390/v6010106.
168. Pecman, A., Kutnjak, D., Gutiérrez-Aguirre, I., Adams, I., Fox, A., Boonham, N., & Ravnikar, M. Next generation sequencing for detection and discovery of plant viruses and viroids: Comparison of two approaches. Front Microbiol 8, 1–10 (2017). Doi: 10.3389/FMICB.2017.01998/FULL.
169. Pappas, N. et al. Virus Bioinformatics. in Encyclopedia of Virology 124–132 (Elsevier, 2021). Doi:10.1016/b978-0-12-814515-9.00034-5.
Received: 28 September 2023/ Accepted: 15 November 2023 / Published:15 December 2023
Citation: Ganchozo-Mendoza, E., Flores, F.J., Garcés-Fiallos, F.R. Virosis en el cultivo de tabaco. Revis Bionatura 2023; 8 (4) 30. http://dx.doi.org/10.21931/RB/2023.08.04.30
Additional information Correspondence should be addressed to felipe.garces@utm.edu.ec



















