Vol 7 No 3 2022- 55

Caracterización fenotípica y molecular de aislamientos clínicos de Saphylococcus aureus resistentes a meticilina obtenidos de dos centros hospitalarios de Tegucigalpa, Honduras
Phenotypic and molecular characterization of clinical isolates of methicillin-resistant Staphylococcus aureus obtained from two hospitals in Tegucigalpa, Honduras
Lilian Sosa 1, Lupe Carolina Espinoza 2, Jhunior Marcia Fuentes 3, María de Lourdes Enríquez de Madrid 4, Norman Rojas Campos 5.
1 Grupo de Investigación en Tecnología Farmacéutica, Facultad de Ciencias Químicas y Farmacia, Universidad Nacional Autónoma de Honduras (UNAH); lilian.sosa@unah.edu.hn
2 Departamento de Química y Ciencias Exactas, Universidad Técnica Particular de Loja (UTPL), Loja, Ecuador; lcespinoza@utpl.edu.ec
3 Faculty of Technological Sciences, National University of Agriculture (UNAG), Honduras. juniorabrahamm@yahoo.com
4 Instituto de Investigaciones en Microbiología, Facultad de Ciencias, Universidad Nacional Autónoma de Honduras (UNAH), Tegucigalpa, Honduras. lourdes@unah.edu.hn
5 Departamento de Bacteriología, Universidad de Costa Rica (UCR), San José, Costa Rica. norman.rojas@ucr.ac.cr
* Correspondence: lilian.sosa@unah.edu.hn; Tel.: +50494505228
Available from: http://dx.doi.org/10.21931/RB/2022.07.03.55
RESUMEN
La aparición de resistencia a los antibióticos por parte de S. aureus se ha convertido en un grave problema de salud pública. Se determinó el porcentaje de S. aureus meticilino-resistente (SARM) en 96 aislamientos clínicos que se encontraban en un banco de bacterias en el laboratorio MEIZ de la Escuela de Microbiología de la UNAH. Las pruebas utilizadas para determinar el porcentaje de SARM fueron mediante difusión en disco utilizando: oxacilina (1µg), meticilina (5µg) y cefoxitina (30 µg). La Concentración Mínima Inhibitoria (CMI) se obtuvo con la prueba de E-test® (Solna, Suecia) para oxacilina. Se detectó la presencia de la proteína PBP2a mediante una prueba de aglutinación en látex y se desarrolló un PCR múltiplex para la detección del gen mecA. Finalmente, se determinó el índice kappa de Cohen, para evaluar la correlación entre las diferentes técnicas utilizadas. El porcentaje de resistencia a oxacilina fue del 15%. Hubo una excelente correlación entre las técnicas. La sensibilidad y especificidad para las diferentes técnicas de detección fue por encima del 90%. De acuerdo al índice Kappa de Cohen, cada uno de los métodos utilizados fueron catalogados como muy buenos para la detección de SARM.
Palabras clave: S. aureus; resistencia fenotípica; SARM; PBP2a, gen MecA.
ABSTRACT
The appearance of antibiotic resistance by S. aureus has become a severe public health problem. Objectives. The percentage of methicillin-resistant S. aureus (MRSA) was determined in 96 clinical isolates found in a bacteria bank at the MEIZ laboratory of the UNAH School of Microbiology. The tests used to determine the percentage of MRSA were by disk diffusion using: oxacillin (1µg), methicillin (5µg) and cefoxitin (30µg). The Minimum Inhibitory Concentration (MIC) was obtained with the E-test® (Solna, Sweden) for oxacillin. The presence of the PBP2a protein was detected by a latex agglutination test, and a multiplex PCR was developed to detect the mecA gene. Finally, Cohen’s kappa index was determined to evaluate the correlation between the techniques used. Results. The percentage of resistance to oxacillin was 15%. There was an excellent correlation between the processes used. The sensitivity and specificity of the different detection methods were above 90%. According to Cohen’s Kappa index, each technique used was very good for detecting MRSA.
Keywords: S. aureus; phenotypic resistance; MRSA; PBP2a, MecA gene.
INTRODUCCIÓN
S. aureus es una bacteria Gram positiva, inmóvil, catalasa y coagulasa positiva; y aunque el género Staphylococcus incluye 52 especies y 28 subespecies, S. aureus es, con mucho, la especie clínicamente más relevante. S. aureus se encuentra en el ser humano como parte de la microbiota comensal de la mucosa nasal y piel de un 20-40% y cuando estas barreras se rompen, por ejemplo, debido a condiciones crónicas de las mismas, heridas o intervención quirúrgica, esta bacteria puede acceder a los tejidos subyacentes o al torrente sanguíneo y causar infección. Personas con dispositivos médicos invasivos (como dispositivos periféricos y centrales), catéteres venosos o sistemas inmunológicos comprometidos son particularmente vulnerables a la infección por S. aureus 1.
Los tratamientos para este tipo de infecciones causadas por esta bacteria pueden variar, los fármacos de primera línea son los antibióticos betaláctamicos tipo penicilina, no obstante, con la aparición inminente de la resistencia microbiana, se han optado por tratamientos como cefalosporinas de quinta generación, macrólidos, fluoroquinolonas, aminoglucósidos y en última instancia los glicopéptidos tipo vancomicina. Sin embargo, y a pesar del gran arsenal terapéutico con el que actualmente se cuenta, ha surgido un grave problema de salud pública como ser la aparición de cepas de S. aureus resistentes a la meticilina conocido por sus siglas como SARM 2.
Las infecciones ocasionadas por SARM, son un problema de salud pública importante en todo el mundo, siendo un número pequeño de clonas las responsables de las infecciones estafilocócicas. El problema actual se presenta sobre todo en los hospitales (SARM-HA), debido al descuido de las técnicas asépticas, hecho que se agrava por la aparición de cepas resistentes a los antimicrobianos, ya que, desde la aparición de los antibióticos, S. aureus ha expresado una excepcional capacidad para producir variantes resistentes. En la pasada década, la epidemiología de las infecciones estafilocócicas cambió particularmente como consecuencia de dos factores principales: el incremento en el número de pacientes externos con excesivo contacto en centros de salud, y la emergencia de clones adquiridos en la comunidad (SARM-CA), por personas sin algún tipo de contacto con Hospitales o centros de salud, hecho que ha causado preocupación entre los profesionales de la salud 3.
La resistencia a antibióticos por parte del S. aureus en especial a los betalactámicos, se debe a un mecanismo de resistencia denominado resistencia intrínseca; también llamado resistencia a la meticilina, el cual es un fenómeno genéticamente complejo donde la cepas de S. aureus normalmente tienen cuatro tipos de proteínas de unión a penicilinas (PBP, por sus siglas en inglés), PBP 1, 2, 3 Y 4, mientras que SARM produce una quinta PBP o “extra” denominada PBP2a o PBP2’, que tiene menor afinidad a la meticilina y al resto de los antibióticos betalactámicos (figura 1). Esta proteína PBP 2a es el producto de la transcripción de un gen denominado mecA, el cual es un segmento de ADN de un tamaño aproximado de 2 Kb que posee dos elementos reguladores (mecR1- mecI) que controlan la trascripción del gen, el cual se encuentra ubicado en un elemento genético denominado Casete Cromosómico Estafilocócico (SCCmec). El SCCmec es un elemento genéticamente móvil, además de contener el gen mecA, contiene un sitio de inserción preferencial para transposones y copias integradas de plásmidos que llevan varios genes de resistencia para antibióticos no betalactámicos. Por lo que, una vez adquirida la resistencia a betalactámicos, existe la potencialidad de que sea acompañada o seguida de la resistencia a otros antibióticos 4.
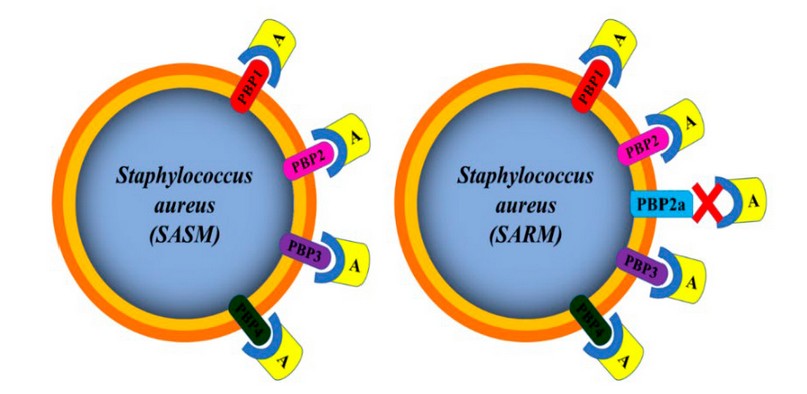
Figura 1. Mecanismo de resistencia por parte del S. aureus a los antibióticos betalactámicos (fuente: creación propia).
A nivel mundial, para el 2016, países como España, Portugal e Italia habían reportado casos de SARM de un 25 a 50%. Francia, Alemania y Reino Unido obtuvieron del 10-25% de casos, países nórdicos como Suecia, Noruega y Finlandia reportaron casos de SARM de 1-5%. En Sudamérica, países como Colombia, Ecuador, Chile y Argentina han obtenido de un 25 a 50%, mientras que Brasil, Venezuela y Perú han alcanzado hasta un 50% de casos de SARM 5.
En Honduras, la situación de SARM ha sido poco estudiada, solamente existen dos trabajos publicados referentes al tema. En primera instancia, tenemos el estudio de Zepeda y col, realizado en 1977 donde se encontró un porcentaje de resistencia a la meticilina del 43%6. Asimismo, el Departamento de Epidemiología del Hospital Escuela (HE), para el año 2011 comunicó un porcentaje de SARM del 28% (Departamento de Epidemiología, HE, Comunicación Oral) y finalmente, Pineda y col para el 2013, manifestaron un porcentaje de resistencia a meticilina del 14%7.
La detección de SARM suele ser en ocasiones compleja debido a la heterogeneidad de su expresión fenotípica, es decir, a pesar de que todas las células de una población posean el gen, solo 1 en 10 la manifiestan, dificultando su detección en el laboratorio con pruebas convencionales, lo que ha llevado al desarrollo y aplicación de diversas técnicas para la detección de cepas SARM mediante la búsqueda de metodologías sensibles y específicas7.
Existen diferentes técnicas para la detección de aislados clínicos de S.aureus que son resistentes a meticilina. Dentro de estas técnicas, se encuentran: prueba de difusión en disco con Cefoxitina, Microdilución en caldo para medir la CMI, Vitek-2, E-test®, pruebas de aglutinación en látex. Asimismo, existen pruebas moleculares para la detección del gen MecA mediante Reacción en Cadena de la Polimerasa (PCR, por sus siglas en inglés)8.
Es por esto que en el presente trabajo se planteó determinar la resistencia fenotípica y molecular a antibióticos betalactámicos de los aislamientos obtenidos del banco de bacterias del Laboratorio MEIZ de la Escuela de Microbiología de la Facultad de Ciencias de la UNAH. Para detectar el porcentaje de resistencia de estos aislamientos, se realizaron una serie de técnicas como son: prueba de difusión en disco con Cefoxitina (FOX), Oxacilina (OX) y Meticilina (Met), detección de la CMI mediante la técnica de Epsilon Test (E-Test®) con tiras de oxacilina (OX), detección de la PBP2a mediante la técnica de aglutinación en látex y la identificación del gen mecA mediante PCR. Asimismo, se hizo una correlación entre las técnicas utilizadas, y finalmente, se determinó la sensibilidad y especificidad de estas técnicas utilizando como ¨gold standard¨ la detección del gen mecA.
MATERIALES Y MÉTODOS
Materiales
Los 96 aislamientos bacterianos de S. aureus obtenidos del banco de bacterias del laboratorio MEIZ, se obtuvieron en el periodo comprendido de octubre del 2010 a junio del 2011 provenientes de pacientes que acudieron al Hospital Escuela (HE) y al Instituto Hondureño de Seguridad Social (IHSS) tanto de consulta externa como del área de hospitalización.
Las cepas utilizadas como controles en todos los experimentos incluyeron: SASM ATCC 29213 (mecA negativo o cepa susceptible) y la SARM CUG36501 (mecA positivo o cepa resistente). La primera cepa fue obtenida del Laboratorio de Bacteriología del Centro de Salud Alonzo Suazo (Tegucigalpa, Honduras) y la segunda cepa fue obtenida del Laboratorio de Bacteriología de la Facultad de Microbiología de la Universidad de Costa Rica (UCR).
Los discos de antibióticos de FOX 30 µg, OX 1 µg y Met 5 µg, se obtuvieron de OxoidTM Thermo Fisher ScientificTM (Waltham, Massachusetts, USA), las tiras de Ox se obtuvieron de E-test® (AB Biodisk, Solna, Suecia). La prueba de aglutinación en látex y los caldos de cultivo para la preparación de Agar simple se obtuvieron de OxoidTM Thermo Fisher ScientificTM ( Waltham, Massachusetts, USA).
Métodos
Identificación de los aislamientos bacterianos
Los aislamientos fueron identificaron nuevamente. Se descongelaron y se cultivaron a 37°C en medio gelosa suplementado con 5% de sangre de carnero, para después ser re-identificadas con la tinción de Gram, prueba de catalasa y se utilizó como ¨Gold standard¨ la prueba de coagulasa en tubo.
Tinción de gram
En un portaobjetos se colocó una gota de agua destilada estéril, y con ayuda de un asa bacteriológica o un cotonete estéril, se tomó una muestra de una colonia de bacteria previamente sembrada en gelosa sangre al 5%, se extendió en el portaobjetos y se combinó con la gota de agua destilada. Una vez que esta se secó, se fijó la muestra con ayuda de metanol. Posteriormente, se agregaron los siguientes reactivos: lugol (1 minuto), mezcla de etanol/acetona (1:1) (4 segundos), safranina (1 minuto). Se lavó con agua hasta que se secó la placa. Finalmente, se agregó aceite de inmersión (1 gota) para llevar al microscopio. Un color morado y cocos dispuestos en grupos (parecido a racimos de uvas) indicaron positividad.
Prueba de catalasa en portaobjetos
En un portaobjetos se colocó una gota de agua destilada y una gota de peróxido de hidrógeno al 3%. Con ayuda de un asa bacteriológica o cotonete estéril, se tomó una colonia de la bacteria previamente sembrada en gelosa sangre al 5% y se combinó con la gota de agua y con la gota de peróxido de hidrogeno a 3%. La presencia de burbujas en el peróxido de hidrogeno indicó positividad mientras que la ausencia de burbujas indicó negatividad.
Prueba de coagulasa en tubo
En un tubo de ensayo se colocó plasma fresco estéril y con ayuda de un asa bacteriológica o cotonete estéril se tomaron de una a dos colonias de la bacteria previamente sembrada en gelosa sangre al 5% y se agitó bien. Se Incubó a 37°C durante 4 horas. La formación de un coágulo indicó positividad. Una vez demostrado que los aislamientos clínicos eran todos S. aureus entonces se procedió a realizar las pruebas de detección de susceptibilidad y/o resistencia a la meticilina.
Prueba de difusión en disco para OX, Met y FOX
Las pruebas de susceptibilidad a OX, Met y FOX se realizaron con discos de 1 µg, 5 µg y 30 μg, respectivamente. Se prepararon inóculos de cada uno de los aislamientos obtenidos de S.aureus a una concentración de 0.5 de la escala de McFarland. Con ayuda de un cotonete estéril se sembró este inóculo sobre placas de agar Mueller-Hinton. Posteriormente, se colocaron con ayuda de una pinza estéril los discos de antibióticos separados unos de otros a una distancia de 20 mm. Las placas se incubaron a 24 h a 37°C y la formación de los halos de inhibición se midieron en mm. Los puntos de corte para OX, Met y FOX fueron los reportados por la CLSI: OX resistente ≤ 11mm, OX susceptible ≥ 13 mm, Met resistente ≤ 10 mm, Met susceptible ≥ 14 mm, FOX resistente ≤ 21 mm y FOX sensible ≥ 22 mm. Para esta prueba se utilizaron las cepas control 9.
Prueba para determinación de la CMI
Se realizó con la prueba de E-test® (Solna, Suecia) utilizándose tiras de OX. Se prepararon inóculos de cada uno de los aislamientos obtenidos de S. aureus a una concentración de 0.5 de la escala de McFarland. Con ayuda de un cotonete estéril se sembró este inóculo sobre placas de agar Mueller-Hinton. Posteriormente, se colocaron con ayuda de una pinza estéril las tiras de oxacilina, las cuales constan de diversas concentraciones del fármaco que van desde [0.016 hasta 256] µg/mL. Las placas se incubaron a 24 h a 37°C y la formación de un halo alrededor de la tira en forma de elipse, específicamente en su parte más angosta, indicó la CMI encontrada. Los puntos de corte para oxacilina fueron los reportados por la CLSI: OX susceptible ≤ 2 µg/mL, OX resistente ≥ 4 µg/mL. Para esta prueba se utilizaron las cepas control 9.
Detección de PBP2a.
Se utilizó la prueba PBP2a detection (oxoid®) que se basa en la aglutinación de partículas de látex sensibilizadas con anticuerpos monoclonales contra PBP2a. Se colocó en un tubo eppendorff de capacidad de 1 ml, 4 gotas del reactivo número uno y con un asa bacteriológica estéril, se tomaron dos o tres colonias aisladas de S. aureus previamente sembradas en gelosa sangre al 5% y se mezclaron hasta formar una suspensión que se observó homogénea, si es posible con ayuda de un vortex. Posteriormente, se colocó la suspensión en una plancha calefactora o en un baño de agua a 100 °C (previamente calentado) durante 3 minutos. Se Dejó enfriar y se añadió una gota del reactivo de extracción dos al tubo y se mezcló bien. Se centrifugó a 3000 rpm con un radio de rotación de 15 cm o a 4500 rpm con un radio de rotación de 4,5 cm. Se Utilizó el sobrenadante para la prueba. Para cada sobrenadante, se marcó uno de los círculos de la tarjeta de la prueba para probarlo con látex de prueba y otro para hacerlo con látex de control. Se añadió una gota del látex de prueba o del látex de control a cada círculo marcado. Se Dispuso de 50 μl del sobrenadante sobre el círculo de prueba y el círculo de control, con cuidado para evitar el sedimento. Se mezcló bien el látex y el sobrenadante de cada círculo con una varilla de mezclado. Se sujetó y movió la tarjeta durante tres minutos y se observó la aglutinación en condiciones de iluminación normales. Finalmente, se registraron los resultados de las reacciones de prueba y de control. Asimismo, para esta prueba se utilizaron las cepas control.
PCR para la detección del gen MecA
Se prepararon suspensiones de 24 horas de cada uno de los 96 aislamientos clínicos obtenidos en este estudio; la extracción de ADN bacteriano, se realizó mediante la técnica descrita por Boom y colaboradores10. Se realizó la PCR con las siguientes secuencias como cebadores: mecA plus 5’-TGG CTA TCG TGT CAC AAT CG-3’ y mecA minus 5’-CTG-GAA-CTT-GTT-GAC-CAG-AG-3. El producto de PCR generado tuvo un tamaño de 310 pb. Cada muestra también se probó con la secuencia de 370 pb del gen ribosomal 16S; los cebadores fueron: 16S plus: 5’-AGG AGG TGA TCC AAC CGC A-3’ y 16S minus: 5’-AAC TGG AAG AAG GTG AT-3’. Asimismo, 2 µL del ADN extraído se agregaron a una mezcla que contenía: 200 µM desoxinucleótidos trifosfato; 10 mM Tris (pH 8.3); 50 mM KCl; 1.5 µM MgCl2; 50 pmol de cebadores y 1.25 U de Taq DNA polimerasa (Promega®).
La amplificación se realizó en un termociclador de acuerdo al siguiente protocolo: desnaturalización a 94°C por 5 minutos, seguido de 30 ciclos a 94°C por 30 segundos, 56°C por 30 segundos y 72°C por 30 segundos, con una extensión final a 72°C por 5 minutos. Los productos amplificados fueron separados mediante electroforesis en gel con agarosa al 2% a 100 V por 30 minutos y teñidos con bromuro de etídio, las bandas fueron visualizadas en un transiluminador con luz ultravioleta.
Análisis estadístico para la correlación de las técnicas
Los datos se tabularon y se analizaron con el programa epidata versión 3.0. Se utilizó el índice Kappa de Cohen como coeficiente de correlación para escalas nominales que midieron la confiabilidad y validez del diagnóstico. Un buen método diagnóstico exhibe buena correlación; los criterios para la interpretación de la fuerza de correlación del índice kappa van de acuerdo con el valor de k: < 0.20 pobre, 0.21-0.40 débil, 0.41-0.60 moderada, 0.61 – 0.80 buena, 0.81- 1.00 muy buena. Se determinó la sensibilidad y especificidad de las técnicas utilizadas.
RESULTADOS
Pruebas de identificación
De los 96 aislamientos clínicos que se obtuvieron del banco de bacterias, todas fueron identificadas nuevamente como S. aureus. Todos los aislamientos fueron gram positivos, catalasa y coagulasa positivo (ver figura 2). Las bacterias se observaron bajo el microscopio como cocos dispuestos en racimos y de color morado. Al momento de poner en contacto la colonia de la bacteria con el peróxido de hidrogeno al 3% este hizo efervescencia al instante. Referente a la prueba de coagulasa, todos los aislamientos clínicos formaron un coagulo de fibrinógeno. Las colonias de las bacterias a simple vista se observaron color dorado, solamente un aislamiento se observó color grisáceo (figura 3).

Figura 2. Pruebas de detección de S. aureus. A. Cocos Gram positivos. B. Prueba de catalasa positiva. C. Prueba de coagulasa en tubo. El tubo de la izquierda muestra una prueba positiva.
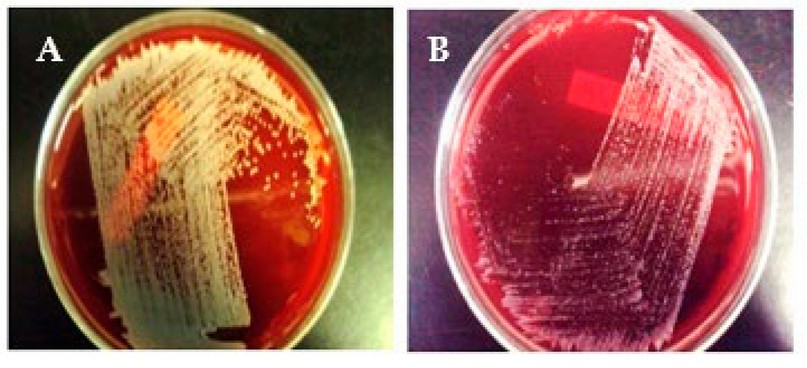
Figura 3. Aislamientos bacterianos de S.aureus. A. aislamiento color dorado. B. Aislamiento color grisáceo.
Prueba de difusión en disco para OX, Met, FOX
En las figuras 4, 5, 6 se muestran las frecuencias de los diámetros de los halos de inhibición obtenidos al enfrentar los discos de OX, Met, FOX con los aislamientos clínicos de S. aureus. Los halos de inhibición con los tres discos de antibióticos fueron menores a los 10 mm de diámetro en 15 de los 96 aislados (15.63%), interpretándose como resistentes. La mayoría de los aislados susceptibles presentaron halos de inhibición entre 13-19 mm para OX y entre 14-19 mm para Met y arriba de los 23 mm para la FOX. No se observaron halos de inhibición que estuvieran dentro del rango intermedio.
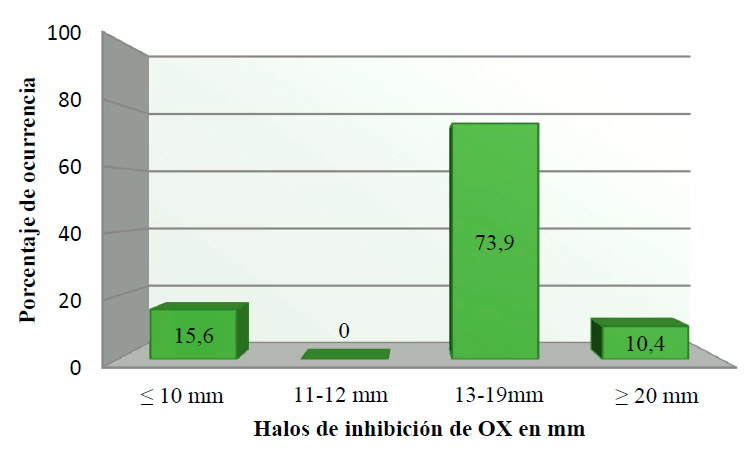
Figura 4. Porcentaje de aislamientos de S. aureus Susceptibles y/o resistentes a la OX.
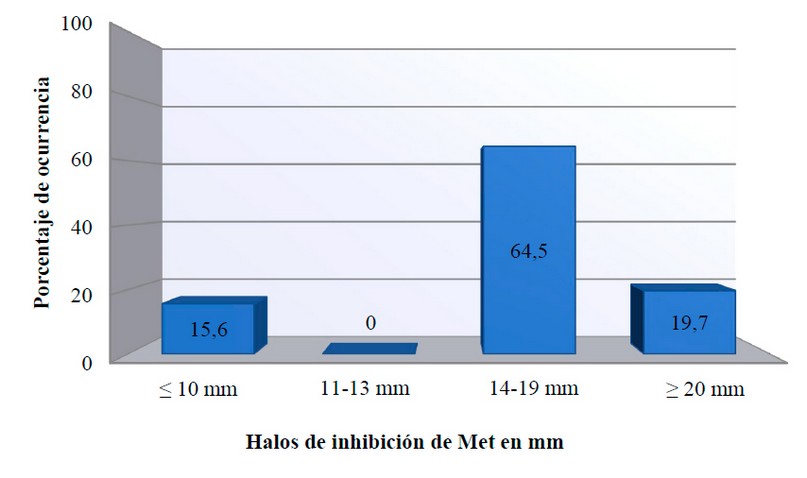
Figura 5. Porcentaje de aislamientos de S. aureus Susceptibles y/o resistentes a la Met.
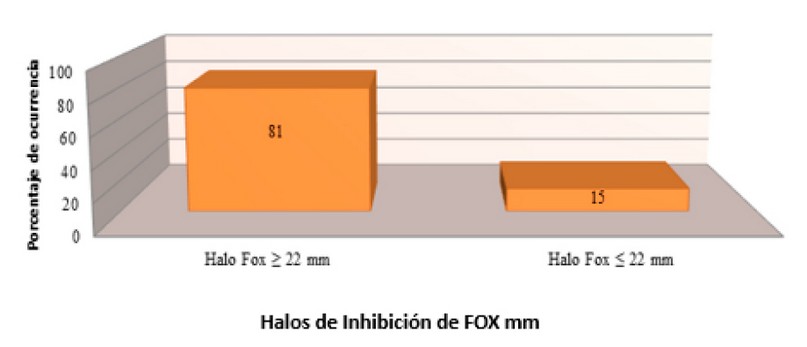
Figura 6. Porcentaje de resistencia de los aislamientos clínicos de S. aureus a la FOX.
Mientras se realizaron las pruebas de difusión en disco, se pudo observar la formación de un ¨pseudohalo¨de inhibición con un crecimiento bacteriano dentro del mismo (figura 7). Para este caso en particular, se tomaron las colonias de bacterias que se encontraban dentro del ¨pseudohalo¨ y se reideintificaron como S. aureus. De esta manera se descartó una contaminación por otras bacterias.

Figura 7. A. Formación de un ¨pseudohalo¨ de inhibición alrededor del disco de OX. B. Halos de inhibición bien formados y delimitados en aislamientos de S. aureus susceptibles a antibióticos.
Prueba para determinación de la CMI
De acuerdo a los puntos de corte establecidos por el CLSI, se consideraron susceptibles a OX aquellos aislamientos con valores de CMI ≤ 2 µg/mL y resistentes si la CMI ≥ 4 µg/mL. En la figura 8 se puede observar la distribución de frecuencias de CMIs de OX que se presentaron en el ensayo de E-test®. El 50% mostró una CMI ≤ 0.38 µg/ml (CMI50) y el 90% una CMI ≤ 32 µg/ml (CMI90). En total mediante esta técnica se detectaron 14 (14.6%) aislamientos con CMIs por encima de los 4 µg/ml, considerados como resistentes, y 82 aislados fueron susceptibles, con una CMI menor a 2 µg/ml (85.4%) (figura 8).

Figura 8. CMI de OX en aislados de S. aureus
La determinación de CMI con oxacilina produjo halos de inhibición elípticos muy bien definidos en todos los aislamientos. Solamente un aislamiento (resistente) formó un “pseudohalo” alrededor de la tira, similar al formado en la prueba de difusión en disco. La CMI encontrada fue de 48 µg/ml, con la aparición de colonias dentro de la elipse por lo que fue clasificado como resistente a oxacilina (figura 9). Por otro lado, se observó un aislamiento que había presentado resistencia en la prueba de difusión en disco, No obstante, esta mostró una CMI de 1.5 µg/mL clasificado como susceptible de acuerdo al CLSI. En vista a lo anteriormente expuesto, es vital la realización de pruebas complementarias. Mediante estas técnicas se determinó un porcentaje de SARM del 14.58%.

Figura 9. Formación de un ¨pseudohalo¨ de inhibición alrededor de la tira de OX.
Detección de PBP2a.
De los 96 aislamientos clínicos incluidos en el estudio, un total de 82 no mostraron ninguna aglutinación mediante la técnica utilizada, lo que se interpretó como ausencia de la PBP2a. 81 aislados fueron susceptibles a Met mediante la prueba fenotípica de difusión en disco. Solamente un aislamiento que mostró resistencia a los betalactámicos, no expresó la proteína PBP2a. Finalmente, 14 fueron positivos por PBP2a, observándose grumos grandes y bien definidos dentro de los primeros 3 minutos (Figura 10).
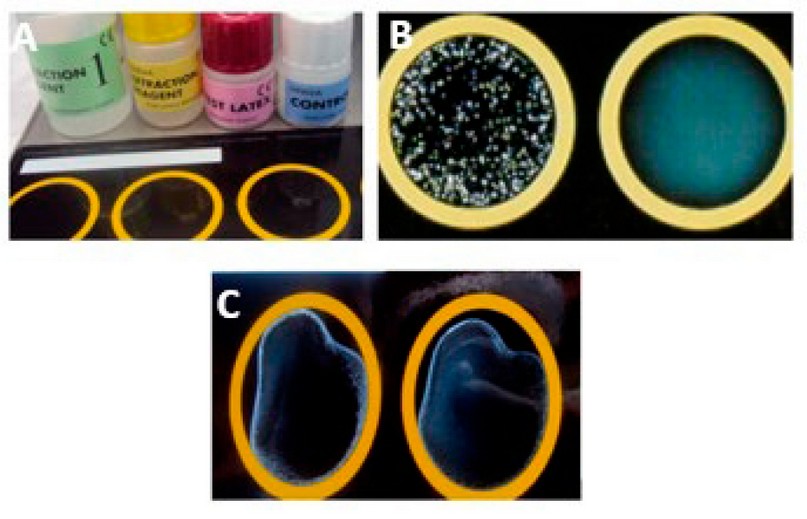
Figura 10. Prueba de aglutinación en látex. A. Kit comercial Oxoid® PBP2a látex; B. Control positivo. C. Aislados positivos para la detección de PBP2a.
Detección del gen mecA
La estandarización del ensayo para la detección molecular del gen mecA no presentó ningún problema, desde la extracción de ADN hasta el desarrollo de una PCR múltiplex la cual detectó simultáneamente 2 regiones altamente conservadas de los genes en estudio. El producto de amplificación fue idéntico en todos los aislamientos mecA positivos, independientemente del nivel de resistencia medido en términos de CMI y difusión en disco.
Todos los aislamientos presentaron las bandas de 370 pb correspondientes al gen ribosómico ARNr 16S, altamente conservado en bacterias. Un total de 14 aislamientos fueron mecA positivos, mostrando bandas de identificación muy bien definidas de 310 pb, como se muestra en la figura 11 y 12. En los pocillos señalados con flechas se colocaron aislados clínicos que fueron fenotípicamente resistentes y mecA positivos.

Figura 11. PCR multiplex de los aislamientos clínicos de S. aureus. M, marcador de peso molecular; C-, control negativo; C+, control positivo. B, blanco. Flechas, aislados positivos por mecA
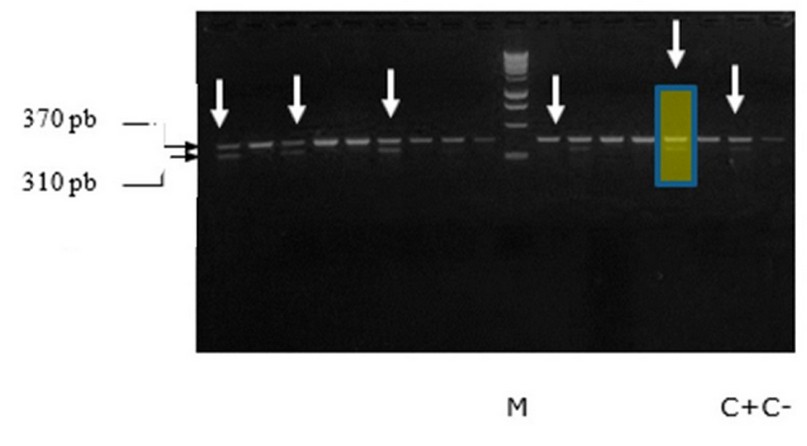
Figura 12. PCR multiplex de los aislados clínicos de S. aureus. M, marcador de peso molecular; C-, control negativo; C+, control positivo. Flechas, aislados positivos por mecA; pocillo señalado y sombreado en amarillo, corresponde a un aislamiento fenotípicamente resistente con el método de difusión en disco y susceptible utilizando las tiras E-test® (con una CMI 1.5 µg/ml) pero que genotípicamente fue resistente.
En la figura 13 se observa que todos los aislamientos fueron mecA negativos. En el pocillo señalado con la flecha se colocó el aislamiento fenotípicamente resistente, pero que no expresó la PBP2a ni el gen mecA.
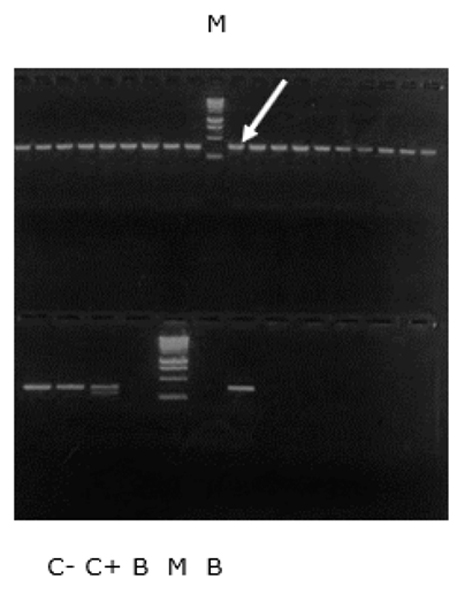
Figura 13. PCR multiplex de los aislados clínicos de S. aureus. M, marcador de peso molecular; C-, control negativo; C+, control positivo; B, blanco; Flecha, aislamiento fenotípicamente resistente pero que no presentó positividad de PBP2a ni presencia de gen mecA.
Análisis estadístico para la comparación de pruebas
Pruebas de concordancia mediante la aplicación del índice Kappa
A continuación, se presentan las pruebas de concordancia entre las diversas técnicas utilizadas en este estudio. En la tabla 1 se resumen los resultados de los índices kappa alcanzados, en donde se mostró una excelente concordancia (95.94%) entre las técnicas de difusión en disco con la detección de CMI mediante E-test®, detección de PBP2a y del gen mecA. Por otro lado, el índice Kappa mostró una excelente concordancia (91.64%) entre las técnicas de E-test® con la detección de la PBP2a y del gen mecA. Finalmente, la concordancia existente entre la expresión de la proteína PBP2a y el gen mecA fue la mejor (100 %).
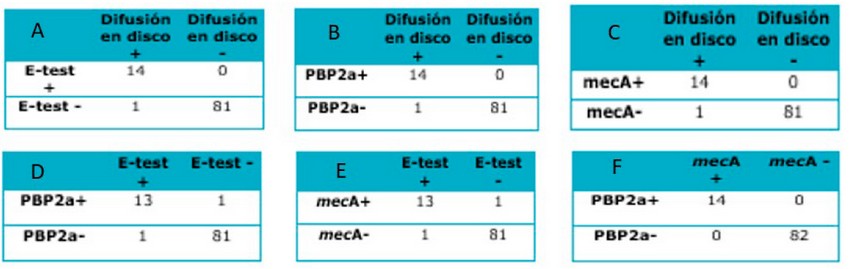
Tabla 1. Concordancia entre las técnicas utilizadas para la detección de SARM*
*A. Correlación entre la prueba de difusión en disco y E-test®. B. Correlación entre la prueba de difusión en disco y la prueba de aglutinación en látex PBP2a. C. Concordancia entre la prueba de difusión en disco y PCR para detección del gen MecA. D. Concordancia entre la prueba de E-test® y la prueba de aglutinación en látex PBP2a. E. Concordancia entre la prueba de E-test® y la PCR para detección del gen MecA. F. Concordancia entre la prueba PCR para detección del gen MecA y la prueba de aglutinación en látex PBP2a.
Sensibilidad y especificidad de las pruebas
Se calculó la sensibilidad y especificidad de las pruebas realizadas, tomando como prueba de referencia la detección del gen mecA (tabla 2).
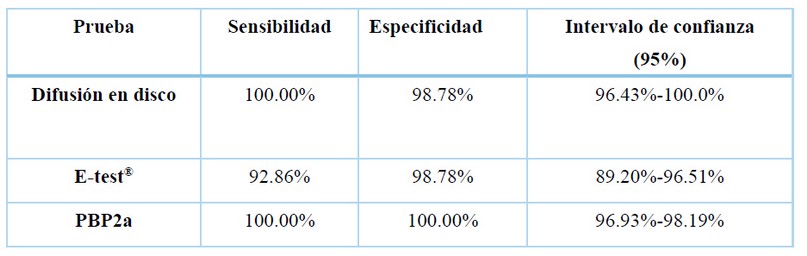
Tabla 2. Sensibilidad y especificidad de las pruebas (IC95%)
DISCUSIÓN
S. aureus es una de las bacterias que se aísla con mayor frecuencia a nivel hospitalario, sin embargo, esta bacteria ha resurgido en la última década como causante de cuadros clínicos muy graves en la comunidad que van desde infecciones de piel y tejidos blandos hasta enfermedades sistémicas que amenazan la vida del paciente. S. aureus es un colonizador habitual de piel y mucosas humanas, así como fómites de los ambientes humanos, tiene la capacidad de evadir la respuesta inmune del hospedero mediante sus múltiples factores de virulencia, como toxinas, enzimas extracelulares y moléculas de superficie, que la convierten en un microorganismo con un alto potencial patogénico11.
S. aureus fue la primera bacteria en desarrollar resistencia a los antibióticos, mediante la producción de una enzima que tenía la capacidad de hidrolizar el anillo betalactámico de la penicilina y de esta forma inactivar la acción farmacológica y terapéutica del antibacteriano. Por esta razón se modificó la estructura química de la penicilina a nivel de grupos sustituyentes como hidroxilo y metilo, dando como resultado la Met, que se convirtió en el tratamiento de elección para tratar las infecciones causadas por los estafilococos. Sin embargo, poco tiempo después de su introducción, se aisló la primera cepa resistente a Met, y desde entonces la incidencia a nivel mundial se ha incrementado progresivamente, por lo que se ha debido recurrir a otras alternativas terapéuticas12.
En este estudio se obtuvieron porcentajes de SARM intermedios (15%) con respecto a los encontrados en años anteriores, en 1977 se publicó un 48% de resistencia, en el año 2011 se comunicó un 28%y para el 2013 se informó un 14%, sin embargo, la muestra obtenida no fue representativa para indicar una prevalencia nacional, sino solamente de una población determinada de la capital. No obstante, estas frecuencias son considerablemente más bajas a las encontradas en otros países del continente americano, y similares a las que presentan algunos países del hemisferio norte. Debido a la ausencia de programas activos de vigilancia y control de brotes de SARM en el país, es importante estudiar a fondo las razones que podrían explicar tales frecuencias, como, por ejemplo, el manejo antimicrobiano de los pacientes, la circulación y disponibilidad de antibióticos a nivel nacional y la detección de portadores de S. aureus 6,7.
La rápida y precisa detección de SARM es importante para guiar una adecuada terapia antibiótica, para esto los laboratorios de microbiología requieren de formación y recursos para identificar correctamente los aislados resistentes. Dentro de las técnicas más utilizadas en Honduras para la determinación de SARM se encuentran el método de difusión en disco con OX y FOX y sistemas automatizados como el Vitek®. La determinación de la CMI mediante microdilución en caldo o por E-test®, y las pruebas como la aglutinación en látex y detección del gen mecA, brindan información complementaria y confirmatoria específica para SARM. En el presente estudio se incorporaron tres de estas técnicas que no suelen utilizarse con frecuencia (pero que son muy sensibles y específicas), con el fin de obtener la identificación definitiva de SARM y poner estas importantes herramientas diagnósticas a disposición del sistema de salud nacional. Sin embargo, estos ensayos son costosos y de difícil adquisición, por lo que la confirmación de SARM requiere una evaluación detallada de la confiabilidad y relación costo-beneficio de las técnicas disponibles 13.
En este estudio se utilizaron dos ensayos de difusión en agar y dos técnicas para la detección de los determinantes de resistencia contra Met, con el fin de evaluar su sensibilidad y especificidad analíticas. Los ensayos con discos o tiras de antibióticos son de fácil implementación y estandarización, ya que los protocolos están ampliamente normalizados y pueden ser aplicados en la mayoría de laboratorios clínicos. Todas las pruebas de control de calidad realizadas para ambas técnicas estuvieron dentro de los parámetros establecidos, lo que validó los resultados obtenidos. No obstante, la correcta identificación de SARM utilizando los métodos convencionales es compleja y algunos aislados son difíciles de clasificar ya que pueden aparecer susceptibles por un método y resistentes o intermedios por otro. Es ampliamente aceptado que la detección de SARM en el laboratorio por métodos de difusión en agar está determinada por varios parámetros como temperatura, periodo de incubación y densidad del inóculo, así como la concentración de sales en los medios de cultivo 14.
En las pruebas de difusión en agar, se presentaron aislamientos SARM con fenotipos de resistencia caracterizados por la formación de halos de inhibición atípicos alrededor del disco de antibiótico, con abundantes colonias intrahalo llamados “halos fantasmas” 15. Uno de ellos también presentó halo fantasma alrededor de la tira E-test®, y fue negativo para PBP2a y gen mecA, pero se consideró como un aislado SARM probablemente con un mecanismo de resistencia distinto a la adquisición plasmídica del SCCmec (gen mecA). Una posibilidad es la producción de un alto nivel de betalactamasas, presente en las cepas conocidas como BORSA16. Alternativamente, este fenotipo de SARM también podría deberse a la modificación de otras PBP tales como PBP1, PBP3 y PBP4 conocidas como cepas MODSA17. En este caso la CMI obtenida para oxacilina indicó un alto nivel de resistencia, más consistente con un fenotipo MODSA, cuya confirmación dependería de pruebas de genotipificación. En estos casos, la correcta identificación de este tipo de resistencia proveería una alternativa terapéutica adecuada.
El otro aislado resistente, resultó positivo para PBP2a y gen mecA, sin embargo presentó una baja CMI por E-test®. Las colonias mostraron diversos tamaños, lo cual se ha descrito en aislados con resistencia heterogénea a los antibióticos. Este tipo de aislados es susceptible a bajas concentraciones de OX y solo una pequeña fracción de la población manifiesta resistencia fenotípica, por lo que su detección a nivel de los laboratorios clínicos puede pasar inadvertida, y facilitar su diseminación intrahospitalaria. De acuerdo con la clasificación de Tomasz, los aislados SARM heterogéneos de clase 1, expresan el gen mecA y poseen una CMI entre 1.5 y 3 µg/ml, lo que concuerda con el fenotipo encontrado en este estudio18. Por consiguiente, para su correcta identificación se requiere de la implementación de pruebas adicionales como aglutinación por látex para PBP2a y PCR.
La técnica de aglutinación en látex para la detección de la PBP2a, fue desarrollada en la década de los 90 con el objetivo de crear una prueba más rápida para la detección de resistencia a Met 19. Desde el año 2002, el CLSI recomendó su uso en caso de no tener técnicas moleculares disponibles, ya que ha demostrado un excelente rendimiento como prueba complementaria para la detección de SARM, destacando su facilidad y rapidez para la obtención de resultados, en comparación con otras metodologías disponibles 9.
En el presente estudio se obtuvo una excelente sensibilidad y especificidad de la prueba de aglutinación en látex. Los resultados mostraron reacciones intensas durante el tiempo establecido y fueron comparables con las cepas control, obteniéndose altos porcentajes de correlación con los métodos implementados en este estudio. Para la interpretación de resultados, se requiere de personal capacitado que puedan brindar resultados óptimos y confiables.
El PCR para la detección del gen mecA ha sido considerado como el método de referencia para la determinación de SARM 20,21. En este estudio se implementó por primera vez en el país esta técnica de diagnóstico molecular para detectar dicho determinante de resistencia a partir de aislados clínicos, y de encontrar posibles cambios en el patrón molecular que se reflejaran en la expresión fenotípica. En otros países del área centroamericana se ha incorporado este ensayo como apoyo diagnóstico y confirmación de la presencia de SARM en muestras clínicas (Cristian Pérez, comunicación personal). El desarrollo de ensayos para la detección de múltiples genes en un solo paso brinda ventajas adicionales como rapidez y validación simultánea de los protocolos de reacción, tal como se observó en este estudio. Los geles de agarosa muestran muy claramente el gen MecA presente en los aislamientos. De estos 15 resistentes fenotípicamente, 14 presentaron el gen.
Todos los métodos utilizados para la detección de resistencia tuvieron una excelente sensibilidad, especificidad, además de una excelente correlación entre técnicas fenotípicas. Por esta razón, es posible sugerir una combinación de al menos dos de estas técnicas para la identificación de aislados SARM. Por ejemplo, el uso del método de difusión en disco con una prueba complementaria como la aglutinación en látex para PBP2a podría ser una buena alternativa diagnóstica para laboratorios con volúmenes pequeños a medianos de muestras positivas por SARM. Asimismo, la combinación de E-test® con la detección del gen mecA provee información más completa de la resistencia a Met, apta para laboratorios de hospitales grandes y centros de referencia nacionales. Además, todas son herramientas valiosas para la adecuada vigilancia epidemiológica, control de la diseminación y de eventuales brotes por este tipo de cepas.
CONCLUSIONES
En este estudio, el porcentaje de resistencia a meticilina por parte de los aislamientos clínicos de S. aureus fue del 15% (mediante la técnica de difusión en disco). No obstante, el porcentaje de resistencia obtenido mediante la prueba de aglutinación en látex para la detección de la PBP2a y el PCR para la identificación del gen MecA fue del 14%. Esto significa que, existen diferentes mecanismos de resistencia por parte de esta bacteria. Específicamente, un caso de SARM presentó resistencia fenotípica pero no genotípica, asimismo, un caso de SARM fue fenotípicamente susceptible por E-test®, pero si presentó resistencia genotípica. Es por esto la importancia de utilizar diferentes técnicas para la detección de cepas SARM ya que esto conlleva al tratamiento oportuno de los pacientes evitando así la aparición de mecanismos de resistencia, específicamente cuando se presentan infecciones causadas por estas cepas con resistencia heterogénea a los betalactámicos. Las pruebas de correlación mostraron que fue conveniente utilizar al menos dos técnicas de identificación de aislamientos SARM. Se observó un fenómeno que hemos denominado como formación de pseudohalos, donde se formó un halo de inhibición alrededor del disco de antibióticos y de la tira de E-test® con crecimiento de S.aureus dentro de los mismos. Todas las técnicas mostraron excelentes porcentajes de sensibilidad y especificidad. Recomendamos realizar este mismo estudio con mayor número de muestras que sea representativa para todo el país. Asimismo, se requieren de estudios de genotipificación para la detección de los SCCmec circulantes en el país para determinar su origen clonal.
REFERENCIAS
1. Becker, K., Schaumburg, F., Fegeler, C., Friedrich, A.W., Köck, R. Prevalence of Multiresistant Microorganisms PMM Study. Staphylococcus aureus from the German general population is highly diverse. Int J Med Microbiol, 2017. 307(1):21-27.
2. Grayson, M. L., Cosgrove, S., Crowe, S. M., Hope, W., McCarthy, J. S., Mills, J., Mouton, J. W., Paterson, D. L. Kucers’ the use of antibiotics: A clinical review of antibacterial, antifungal, antiparasitic, and antiviral drugs, seventh edition. In Kucers the Use of Antibiotics: A Clinical Review of Antibacterial, Antifungal, Antiparasitic, and Antiviral Drugs, Seventh Edition, 2017. (pp. 1-4841).
3. Falcone, M., Serra, P., Venditti, M. Serious infections due to methicillin-resistant Staphylococcus aureus: an evolving challenge for physicians. Eur J Intern Med, 2009, 20(4), 343-7.
4. Fishovitz, J., Hermoso, J.A., Chang, M., Mobashery, S. Penicillin-binding protein 2a of methicillin-resistant Staphylococcus aureus. IUBMB Life, 2014, 66(8), 572-7.
5. Lee, A.S, de Lencastre, H., Garau, J., Kluytmans, J., Malhotra-Kumar, S., Peschel, A., Harbarth, S. Methicillin-resistant Staphylococcus aureus. Nat Rev Dis Primers, 2018, 31, (4),18033.
6. Sosa Díaz, L.E. Caracterización fenotípica y molecular de las cepas de Staphylococcus aureus resistentes a drogas tomadas de pacientes que acudieron al Hospital Escuela y al Instituto Hondureño de Seguridad Social en el período de octubre del 2010 a enero del 2011. (Tesis de máster). Universidad Nacional Autónoma de Honduras (UNAH). 29 de agosto del 2012.
7. Pineda-García, L., Tzoc, E., Herrera, L., Galindo, C., Rivera, M.F. S. aureus y resistencia antimicrobiana en dos hospitales de Tegucigalpa, 2011-2012, Rev. Ciencia y Tecnología, 2013, 13, 30-40.
8. Desta, K., Aklillu, E., Gebrehiwot, Y., Enquselassie, F., Cantillon, D., Al-Hassan, L., Price, J.R, Newport, M.J, Davey, G., Woldeamanuel, Y. High Levels of Methicillin-Resistant Staphylococcus aureus Carriage Among Healthcare Workers at a Teaching Hospital in Addis Ababa Ethiopia: First Evidence Using mecA Detection. Infect Drug Resist, 2022, 17(15), 3135-3147.
9. Clinical and Laboratory Standards Institute (CLSI). Reference method for broth dilution antifungal susceptibility testing of filamentous Fungi: Approved Standard–Second Edition. CLSI Document M38 A2. Wayne, PA: Clinical and Laboratory Standards Institute; 2022.
10. Boom, R., Sol, C.J., Salimans, M.M., Jansen, C.L., Wertheim-van Dillen, P.M., van der Noordaa, J. Rapid and simple method for purification of nucleic acids. J Clin Microbiol, 1990, 28(3):495-503.
11. Aires de Sousa, M., de Lencastre, H. Bridges from hospitals to the laboratory. FEMS Immunol Med Microbiol, 2004, 40:101-111.
12. Woerther, P.L., Burdet, C., Chachaty, E., Andremont, A. Trends in human fecal carriage of extended-spectrum beta-lactamases in the community: Toward the globalization of CTX-M. Clin Microbiol Rev, 2013, 26, pp. 744-758.
13. Maugeri, G., Lychko, I., Sobral, R., Roque, A.C.A. Identification and antibiotic-susceptibility profiling of infectious bacterial agents: a review of current and future trends. Biotechnol. J., 2019, 14(1), e1700750.
14. Kloos, W.E. BT. Staphylococcus and micrococcus in: Murray PR, Baron EJ, Pfaller MA, Tenover FC, Yolken RH (eds) Manual of clinical microbiology. American Society for Microbiology. Washington, DC. 1999, p 264–282.
15. Efectos del los microorganismos, disponile en: https://www.biomerieux.es/sites/subsidiary_es/files/1012_002es99149a_etest_guia_de_lectura_bacterias_aerobias.pdf (accedido en 20 de abril de 2022).
16. Hryniewicz MM, Garbacz K. Borderline oxacillin-resistant Staphylococcus aureus (BORSA) – a more common problem than expected? J Med Microbiol, 2017, 66(10):1367-1373.
17. Gitman, M.R., Alburquerque, B., Chung, M., van de Guchte, A., Sullivan, J.M., Obla, A., Polanco, J., Oussenko, I., Smith, M.L., Samaroo, F., Barackman, D., Altman, D.R., Sordillo, E., van Bakel, H. Modified methicillin-resistant Staphylococcus aureus detected in neonatal intensive care patients, J. Antimicrob. Chemoter, 2021, 76(11), 2774–277.
18. Marlowe, E.M., Cohen, M.D., Hindler, J.F., Ward, K.W., Bruckner, D.A. Practical strategies for detecting and confirming vancomycin-intermediate Staphylococcus aureus: a tertiary-care hospital laboratory’s experience. J Clin Microbiol., 2001, 39(7):2637-9.
19. Van Griethuysen, A., Pouw, M., van Leeuwen, N., Heck, M., Willemse, P., Buiting, A., Kluytmans, J. Rapid slide latex agglutination test for detection of methicillin resistance in Staphylococcus aureus. J Clin Microbiol, 1999, 37(9), 2789-92.
20. Chambers, H.F. Methicillin resistance in staphylococci: molecular and biochemical basis and clinical implications. Clin Microbiol Rev., 1997, 10(4), 781-91.
21. Pournajaf, A., Ardebili, A., Goudarzi, L., Khodabandeh, M., Narimani, T., Abbaszadeh, H. PCR-based identification of methicillin-resistant Staphylococcus aureus strains and their antibiotic resistance profiles. Asian Pac J Trop Biomed, 2014, 4(Suppl 1): S293-7.
Received: 10 April 2022 / Accepted: 12 July 2022 / Published:15 August 2022
Citation: Sosa L, Espinoza L C, Marcia Fuentes J, Enríquez de Madrid M L, Rojas Campos N. Caracterización fenotípica y molecular de aislamientos clínicos de staphylococcus aureus resistentes a meticilina obtenidos de dos centros hospitalarios de Tegucigalpa, Hondura. Revis Bionatura 2022;7(3) 55. http://dx.doi.org/10.21931/RB/2022.07.03.55



















