Vol 7 No 3 2022- 19

Predicción bioinformática de proteínas NBS-LRR en el genoma de Coffea arabica.
Bioinformatic prediction of NBS-LRR proteins in the Coffea arabica genome
Marcela María Moncada1, Manuel Antonio Elvir1, Juan Rafael Lopez3, and Andrés S. Ortiz1, 2*
1 Universidad Nacional Autónoma de Honduras; aortizm@unah.edu.hn
2 Instituto de Investigaciones en Microbiología; aortizm@unah.edu.hn
3 Instituto Hondureño del Café (IHCAFE); juralopez@gmail.co
* Correspondencia: aortizm@unah.edu.hn; Teléfono: (504) 3353-7765
Available from: http://dx.doi.org/10.21931/RB/2022.07.03.19
RESUMEN
Gracias al acceso al genoma completo de Coffea arabica y el Desarrollo de multiples herramientas de bioinformartica que permite la búsqueda de genes de resistencia de plantas (R-genes), ha sido posible implementar estas estrategias en programas de mejora genética. En las plantas, los R-genes codifican proteínas involucradas en mecanismos de defensa contra patógenos. Los genes con dominios tipo Nucleotide-Binding-Site Leucine-Rich-Repeat (NBS-LRR) forman la familia de R-genes de plantas más grande. El objetivo de este estudio fue identificar genes de proteínas NBS-LRR en el genoma de C. arabica utilizando un enfoque bioinformático. Identificamos motivos conservados de R-genes de C. arabica relacionados con genes similares encontrados en Coffea canephora y Coffea eugenoides, dos especies evolutivas relacionadas con C. arabica. Los resultados de estos análisis revelaron proteínas con origen evolutivo provenientes de dicotiledóneo ancestrales, así como proteínas de resistencia específicas del género Coffea. Además, todas las secuencias de los R-genes de C. arabica mostraron una gran similitud con proteína CNL de Arabidopsis thaliana. Finalmente, la presencia de motivos altamente conservados, la distribución cromosómica y las relaciones filogenéticas de los R-genes de C. arabica muestran procesos de coevolución con patógenos adaptados, demostrando de esta manera la importancia del estudio de estos genes en la inmunidad del café.
Palabras clave: Café, NBS-LRR, Proteínas de Resistencia, Bioinformática.
ABSTRACT
Thanks to access to the complete genome of Coffea arabica and the development of multiple bioinformatics tools that allow the search for plant resistance genes (R-genes), it has been possible to implement these strategies in breeding programs. In plants, R-genes encode proteins involved in defense mechanisms against pathogens. Genes with Nucleotide-Binding-Site Leucine-Rich-Repeat (NBS-LRR)-like domains form the most significant plant R-gene family. This study aimed to identify NBS-LRR protein genes in the C. arabica genome using a bioinformatics approach. We identified conserved motifs of C. arabica R-genes related to similar genes found in Coffea canephora and Coffea eugenoides, two evolutionary species related to C. arabica. These analyses revealed proteins with evolutionary origin from ancestral dicotyledons and resistance proteins specific to the genus Coffea. In addition, all the sequences of the C. arabica R-genes showed a high similarity with CNL protein from Ara-bidopsis thaliana. Finally, the presence of highly conserved motifs, chromosomal distribution and phylogenetic relationships of the C. arabica R-genes show coevolution processes with adapted pathogens, thus demonstrating the importance of studying these genes in coffee immunity.
Keywords: Coffee, NBS-LRR, Resistance Proteins, Bioinformatics.
INTRODUCCIÓN
El café es un producto básico importante y una fuente de ingresos para más de 60 países de todo el mundo. El género Coffea contiene más de 90 especies, siendo Coffea arabica y Coffea canephora las especies de mayor valor comercial. La producción de estas especies se distribuye en aproximadamente un 60 y 40% respectivamente (http://www.ico.org/). Coffea arabica es la única especie tetraploide de su género, resultado de la especiación producta de la hibridación de C. canephora y C. eugenioides 1. Coffea arabica es susceptible a varios patógenos, como bacterias, hongos, nematodos e insectos, siendo Hemileia vastatrix e Hypotenemus hampei las principales amenazas para el cultivo 2. Las plantas y los patógenos han desarrollado una interacción altamente adaptada. Por ejemplo, los patógenos biotróficos están mediados por la interacción de genes de resistencia (R-genes) en plantas y genes de avirulencia equivalentes en los patógenos 3,4. Las proteínas de tipo Nucleotide Binding Site-Leucine Rich Repeats (NBS-LRR) son una de las familias de R- genes más importantes. Estas proteínas se consideran de origen antiguo y altamente evolucionadas, ya que pueden interactuar con las proteínas efectoras del patógeno 3–5. Esta familia de proteínas se clasifica en dos grupos según el dominio N-terminal. El primer grupo presenta un dominio de proteína similar al receptor Toll/Interleukin 1 o TIR-NBS-LRR (TNL). El otro grupo contiene un dominio Coiled Coil en la región N-terminal o CC-NBS-LRR (CNL). Ambos grupos están relacionados con la señalización de las apoptosis o reacciones de hipersensibilidad (HR) 5–7.
El dominio NBS de estas proteinas se ha relacionado con motivos altamente conservados en la familia de ATPasas y la proteína G. Motivos como P-loop, RNBS-A, RNBS-B, RNBS-C, RNBS-D, kinase-2, kinase-3a y GLPL, se han identificado en TNL y CNL de plantas. Estos motivos están relacionados con la capacidad del dominio NBS para hidrolizar ATP o GTP, una activación molecular de las proteinas NBS-LRR 5,8. El dominio LRR está compuesto por repeticiones en tándem entre 20 y 30 secuencias consenso tipo LxxLxLxxNxL, donde «L» representa residuos de leucina, «N» puede representar residuos de asparagina, treonina, serina o cisteína, y «x» puede ser cualquier aminoácido. Este dominio se caracteriza por una estructura cóncava formada por una serie de hojas β. Esta región ha sido identificada como responsable de la interacción proteína-proteína 5,8,9. La presencia de NBS-LRR se ha demostrado en todas las especies de plantas; sin embargo, también se observó que las plantas monocotiledóneas no tenían TNL 10,11. A través de estudios moleculares e in sillico, se ha demostrado la presencia de todas las familias de R- genes en Coffea arabica, pero no así hasta este studio en C. arabica 12,13. Se han identificado nueve genes de resistencia a la roya del café (SH1-SH9) mediante el uso de plantas diferenciadoras 14. El gen SH3 ha mostrado dotar de resistencia vertical contra Hemileia vastatrix, este gen ha sido descrito como un gen que codifica una proteína tipo CNL, siendo este gen un elemento importante utilizado en los programas de mejoramiento genético del café a nivel mundial 15.
La identificación y caracterización de R- genes de plantas se ha realizado con enfoques tanto moleculares, como bioinformáticos. Sekhwal (2015) propuso un enfoque que permite identificar análogos de R- genes en genomas de plantas. Este enfoque requiere la generación de bases de datos de referencia de R- genes, informados que puede ser seleccionados a partir de bases de datos públicas especializadas. Posteriormente, estas bases de datos son utilizadas para realizar alineamiento contra el genoma de la planta de interés. Los resultados obtenidos a partir de los alineamientos se someten a procesos de identificación de dominios y motivos conservados que permiten clasificar las secuencias en diferentes familias de R- genes 16. Actualmente, las herramientas bioinformáticas permiten una amplia variedad de diferentes estudios y análisis que predicen con precisión la función y estructura de las proteínas, lo que permite identificar y caracterizaR-genes y proteínas de resistencia de plantas. En este estudio, utilizamos un enfoque bioinformático para identificar secuencias candidatas para NSB-LRR en el genoma de Coffea arabica, así como, para predecir la ubicación, estructura y localización cromosómica de las secuencias identificadas. Permitiendo reconocer nuevos factores de resistencia a enfermedades que puedan ser utilizadas en programas de mejoramiento genético del café.
MATERIALES AND MÉTODOS
Minera de datos
Para identificar los genes de resistencia (R- genes) en el genoma de C. arabica, utilizamos un enfoque descrito por Sekhwal y col. con algunas modificaciones [16]. Los genes codificantes de referencia del Nucleotide Binding Site-Leucine Rich Repeats (NBS-LRR) se recopilaron mediante búsqueda por palabra clave en la base de datos Plant Resistance Genes Database 3.0 17 (PRGdb http://prgdb.org/prgdb/) usando palabras clave como TNL o CNL. Los resultados se almacenaron en archivos fasta y se sometieron a análisis en la versión web de Pfam v.32.0 18 (https://pfam.xfam.org/) para identificar dominios de tipo Toll/Interleukin-like Receptor (TIR), Coiled-Coil (CC), así como, dominios NBS confirmando la familia a la pertenecian los genes de referencia. Las secuencias resultantes se clasificaron en dos grupos en función del dominio N-terminal, TNL para proteínas de referencia que contenían dominios TIR-NBS y CNL para proteínas de referencia con dominios Coiled Coil-NBS. Las secuencias de genes de referencia sin dominios TIR o CC se descartaron de los análisis posteriores. Finalmente, el genoma de Coffea arabica var. Red Bourbon se obtuvo a nivel de scaffolds del sitio web de World Coffee Research (WCR) (https://worldcoffeeresearch.org/work/coffea-arabica-genome/) y se reservó para análisis posteriores.
Predicción de proteínas NBS-LRR de Coffea arabica
Las secuencias del genoma de Coffea arabica se alinearon con los genes CNL y TNL de referencia utilizando el software BLAST versión 2.5.0 para linux (https://ftp.ncbi.nlm.nih.gov/blast/executables/blast+/LATEST/), utilizando los parámetros predeterminados excepto el e-value, el cual se estableció en 1×10-14 con el objetivo de evitar alinear secuencias con baja identidad 19. Los resultados se filtraron mediante un script “hecho en casa” para el paquete seqinr v.3.6-1 R para eliminar redundancias. Posteriormente, las secuencias filtrados del genoma de café, se tradujeron a los seis ORF utilizando la herramienta de traducción ExPASy 20 (https://web.expasy.org/translate/). Las regiones de codificación se extrajeron y sometieron a anotación funcional con el software INTERPROSCAN v.5.39-77.0 21 (http://www.ebi.ac.uk/interpro/search/sequence/). Las secuencias de C. arabica que presentaban anotaciones relacionadas con los dominios de proteína tales como CC, TIR, NBS y LRR se recuperaron para usarlas en los análisis posteriores. Despues, y debido a que solo las proteínas de tipo CNL de C. arabica (CNL-CA) presentaron los tres dominios clásicos de las NBS-LRR fueron seleccionadas para los analisis subsiguientes. Más tardes, las secuencias de tipo CNL de C. arabica (CNL-CA) se sometieron a análisis de identificación de motivos en el software MEME suite v5.1.1 ejecutable para Linux 22 (http://meme-suite.org/) se utilizaron parámetros predeterminados, modificando la búsqueda de motivos a un máximo de 45 motivos. Las secuencias fueron luego agrupadas según los patrones de motivos compartidos, gracias a la alta similitud (> 98%) entre las secuencias. Las secuencias agrupadas por patrones de motivos, fueron utilizadas en los análisis posteriores.
Análisis filogenético
Los CNL-CA se alinearon con y sin las secuencias de referencia de plantas con el algoritmo MUSCLE en el software AliView para Linux (https://ormbunkar.se/aliview/) para calcular árboles filogenéticos entre secuencias CNL-CA y entre estas y secuencias de referencia. Utilizamos RAxML del servidor CIPRES Science Gateway v.3.3. (https://www.phylo.org/), empleando el método de Máxima Verosimilitud, con 1000 bootstraps, el modelo evolutivo Jones-Taylor-Thornton, con estimación de sitios invariantes, y distribución gamma (JTT+G+I).
Ubicación cromosómica
Para predecir la ubicación cromosómica, los CNL-CA se alinearon con pseudocromosomas de Coffea canephora, una especie estrechamente relacionada con C. arabica. Utilizamos el software de alineamiento del sitio web Coffee Genome Hub 23 (http://coffee-genome.org/) con un e-value de 1×10-100 y un máximo de 20 secuencias de salida. Luego, los resultados fueron filtrados eliminando secuencias con menos del 85% de similitud y cobertura.
Predicción estructural.
El plegamiento de proteínas de los CNL-CA se predijo sometiendo una secuencia representative de cada grupo de patornes de motivos de CNL-CA en el software Phyre v2.0 24 (http://www.sbg.bio.ic.ac.uk/~phyre2/html/page.cgi?id= índice) con parámetros predeterminados. Simultáneamente, el peso molecular y el punto isoeléctrico se calcularon el sitio web ExPasy Compute pI/Mw 20 (https://web.expasy.org/compute_pi/) utilizando los parámetros predeterminados.
RESULTS
Data Mining
Se recuperaron un total de 153 secuencias de la base de datos PRGdb a través de la estrategia de palabras clave. Las anotaciones de secuencia confirmaron la presencia de dominios de proteína NBS-LRR. Se seleccionaron 53 secuencias que presentaban dominios relacionados con secuencias tipo CNL y 17 con secuencias tipo TNL. Posteriormente estas fueron utilizadas como secuencias de referencia en el alineamiento con las secuencias del genoma de Coffea arabica. También se recuperamos 164,254 secuencias a nivel de scaffolds del genoma completo de WCR de Coffea arabica.
Predicción de proteínas NBS-LRR
Se identificaron un total de 4519 secuencias de tipo CNL y 1983 de tipo TNL en el genoma del C. arabica. Posteriormente, se eliminaron los resultados redundantes, la secuencias resultantes fueron sometidas a anotación functional. Se identificaron y anotaron un total de 76 proteínas tipo NBS-LRR, distribuidos en 35 CNL (46 %), 19 TNL y 41 secuencias (29%) que solo contenian uno o dos de los dominios típicos de las proteínas NBS-LRR. Finalmente, solo las secuencias tipo CNL mostrarón los tres dominios de la proteína en el mismo ORF (Tabla complementaria 1). Las anotaciones más frecuentes dentro de los CNL-CA fueron: dominios NB-ARC (IPR002182), dominio Rx N-Terminal (IPR041118), superfamilia de dominios Leucine Rich Repeat (IPR03675), dominios Virus X resistance protein-like, coiled coil domains (IPR038005), P-loop contenidos en dominios de tipo nucleótido trifosfato hidrolasa (IPR027417), así como, superfamilia de dominios Winged helix-like DNA binding (IPR03388). Todas las anotaciones generadas fueron relacionadas con los dominios de proteína tipo CNL.
Se identificaron dieciséis motivos altamente conservados en las secuencias de CNL-CA. Tres de estos motivos se localizaron en el dominio CC, cuatro en el NBS y nueve en los dominios LRR (tabla 1). Todos los motivos altamente conservados mostraron similitud con las proteínas de resistencia de Coffea arabica, C. canephora o C. eugenioides disponibles en las base de datos del NCBI. Ademas, cuatro de los 16 motivos mostraron similitudes con proteínas de resistencia de plantas que no pertenecen al género Coffea. El motivo número 5 de las secuencias CNL-CA mostró un 81% de similitud con una proteína de resistencia a enfermedades (DRP) de Corchorus capsularis (Acceso: OMO 94926.1). El motivo número 11, posee un 66% de similaridad a una DRP de Actinidia chinesis (Accesión: PSS24536.1). El motivo número 15 mostró un 64 % de similitud con la proteinas ATPP2-A2 de Hibiscus syriacus (Accesión: KAE8684719.1) mientras que el motivo número 16 es 52 % similar a una DRP putativa de Morella rubra (Accesión: KAB1213552.1). Todas las secuencias pertenecían a proteínas de tipo CNL, aunque ninguno de los motivos que mostraban alta similaridad se ubicaron en la región CC de las proteínas. También fue posible identificar siete motivos conservados relacionados con motivos de tipo P-Loop, RNBS-A, EDVL, GLPL, RNBS-D, Kinase-2 y Kinas-3, los cuales encuentran comunmente en dominios de proteinas NBS. Tambien se identificaron siete patrones de motivos relacionados con el dominio CC (Figura 1). Debido a la gran similitud (> 98 %) entre las secuencias que comparten el mismo patron de motivo, creamos 9 grupos que contenían de 2 a 4 secuencias con patrones altamente consevados cada uno (Tabla 2). Por otro lado, 13 secuencias no mostraron suficiente similitud con otras CNL-CA por lo que, posteriormente, fueron analizadas individualmente (Figura 2).
Análisis filogenético
El árbol filogenético de CNL-CA mostró la ptresencia de cuatro grupos principales. La secuencia Ca_3_358_ORF_2_1 fue segregada del resto de las secuencias y no mostró relación filogenética directa con ninguna otra secuencia CNL-CA (figura 3). El árbol filogenético CNL-CA con secuencias de referencia, reveló cinco grupos diferentes y cuatro secuencias segregadas. Los CNL-CA se ubicaron exclusivamente dentro de los grupos resaltados en color azul y rojo en la figura 4. Los grupos resaltados en amarillo y verde incluían secuencias de plantas monocotiledóneas exclusivamente. El grupo azul incluye unicamente secuencias de plantas dicotiledóneas, mientras que el grupo rojo combina secuencias de dicotiledoneos y monocotiledoneas. La mayoría de las secuencias CNL-CA presentaron algún nivel de relación filogenética con las secuencias de referencia. La secuencia Ca_3_358_ORF_2_1 mostró una relación directa con 9 secuencias de referencia. Por otro lado, la secuencia Ca_50_488_ORF_2_1 no mostró relación filogenética con ninguna otra secuencia de referencia.
Ubicación cromosómica
Todas las secuencias de CNL-CA se alinearon con pseudocromosomas de Coffea canephora. Los pseudocromosomas 3, 4, 5 y 8 mostraron el mayor número de loci alineados con CNL-CA (41, 33, 43 y 51 respectivamente) (Tabla 3). Los alineamientos con los pseudocromosomas 9 y 10 no cumplían los parámetros mínimos de cobertura o identidad establecidos, por lo que fueron omitidos (tabla complementaria 2 Localizacion Cromosómica). Los pseudocromosomas 3 y 8 mostraron el mayor número de CNL-CA sin relación ancestral con genes del núcleo cromosómico de las eudicotiledóneas (Figura 5). Finalmente, se localizaron cuatro copias de la secuencia Ca_50_488_ORF2_1 en el pseudocromosoma 8 de C. arabica, siende este especialemente relevante debido a la falta de relacion filogenetica con otros genes tanto de café, así como de referencia.
Predicción estructural
Para obtener información structural de los genes CNL-CA identificado, se sometimos a analisis de predicción estructural, obteniendo en todos los casos un 100% de confianza y una cobertura de entre 58 al 98% en relación a las proteínas utilizadas como modelo estructural por el software Phyre 2 (tabla 3). También calculamos los pesos moleculares teóricos del CNL-CA, los resultados de este análisis oscilaron entre 78,14 kDa (Group_5_consensus) y 169,39 kDa (Ca_3_358_ORF_2_1). Además, calculamos el punto isoeléctrico teórico de los CNL-CA, el cual resulto entre 5.46 (Ca_75_74_ORF_5_1) a 8.64 (Ca_453_111_ORF_5_1) (tabla 3). Todas las predicciones mostraron una similitud estructural con la Proteína 4 similar a RPP13 de Resistencia a Enfermedades (Accesión: EMD-0680) de Arabidopsis thaliana, las cual es una proteína similar a CNL (Figura 6).
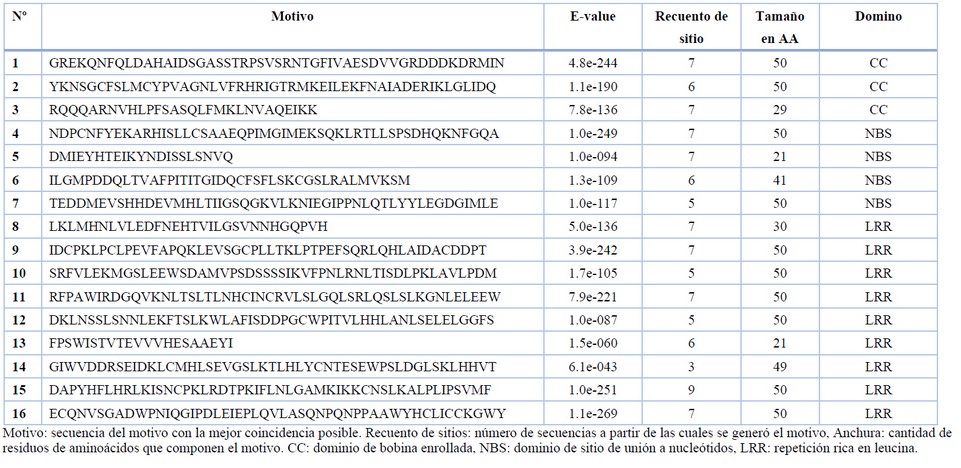
Tabla 1. Características de los motivos conservados de CNL-CA

Tabla 2. Identificaciones de secuencias agrupadas basadas en patrones de motivos

Tabla 3. Características y localización cromosómica de los CNL-PCP

Figura 1. Motivo conservado del dominio NBS identificado en las secuencias CNL-CA.

Figura 2. (a) Patrones de motivos de CNL-CA, cada bloque de color representa un motivo diferente, la longitud del bloque indica el número de residuos de aminoácidos que lo conforman, y la altura de los bloques indica la frecuencia y el grado de coincidencia de los motivos en las secuencias análisadas; (b) Bloques de motivos con su secuencia consenso
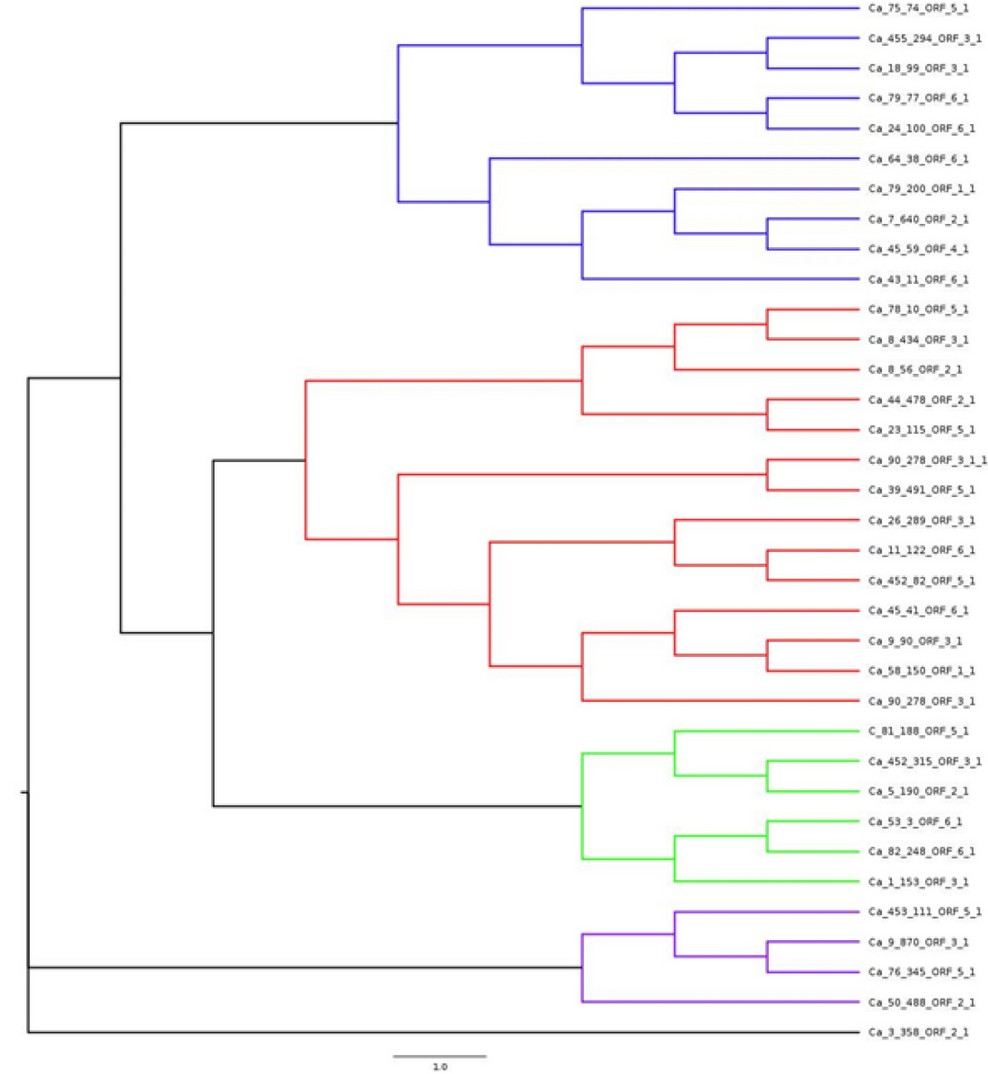
Figura 3. Análisis de máxima verosimilitud de C. arabica CNL.

Figura 4. Análisis de máxima verosimilitud de Coffea arabica y CNL de referencia.

Figura 5. Ubicaciones cromosómica CNL-CA, las regiones de colores indican correspondencia con los 7 bloques ancestral de cromosomas de eudicotiledóneos; las regiones blancas representan regiones cromosómicas del género Coffea; las líneas rosa indican la ubicación de las copias de CNL-CA en los pseudocromosomas de C. canephora.

Figura 6. (a) Estructura en cartoon de la proteína 4 similar a RPP13 de Arabidopsis thaliana (b), (c) y (d) estructuras en cartoon animados de CNL-CA: Consensus 5, Ca_79_200_ORF_1_ y Ca_8_56_ORF_2_1 respectivamente, se muestran hélices alfa en rojo, hojas beta en amarillo y bucles en verde.
DISCUSIÓN
Las proteínas NBS-LRR son factores de resistencia de las plantas contra las enfermedades 3,8. Los CNL y TNL son las subclases de R-genes más diversas, aunque la cantidad de estos factores y la distribución cromosómica varía según la especie de planta 25,26. Identificamos 35 secuencias CNL completas y 19 secuencias TNL truncadas en una proporción de 8:1, similar a la observado en otros estudios relacionados con plantas leguminosas, solanáceas, álamos y vides 10,11,26–29. Sin embargo, se observaron resultados diferentes en especies de la familia Brassicaceae como Arabidopsis thaliana, Arabidopsis lyrata y Brassica rapa, donde la cantidad de TNL fue mayor que la cantidad de CNL, lo que podria resultar de procesos de adaptación a patógenos 11,30. Como sugiere Noir (2002), la presencia de TNL truncado de Coffea arabica podría ser un resultado de procesos de domesticación de la planta, como se ha onservado en otros cultivos 12. Además, se ha observado que las gramíneas así como otros monocotiledones no poseen genes TNL, lo que sugiere que estos se desarrollaron después de la aparición de los monocotiledones 11,29,31. Los CNL se han reportado previamente como importantes factores de resistencia a los patógenos en Coffea arabica. Por ejemplo, el gen SH3 confiere resistencia vertical a Hemileia vastatrix y a su vez este gen codifica una proteina similar a CNL15.
El dominio LRR de los CNL-CA mostró el mayor número de motivos conservados. Sin embargo, no se observaron distribuidos uniformemente entre las secuencias, de manera similar a lo observado en Solanum pimpinellifolium y Arabidopsis thaliana, donde la proporción de motivos conservados de LRR fue mayor que la de NBS y CC 9,32. Esto podría estar relacionado con la estructura terciaria de este dominio y no con la región de interacción proteína-proteína, que es específica para cada proteína de resistencia. El dominio NBS de los CNL-CA presentó siete motivos conservados diferentes, todos ellos previamente informados en R-genes de plantas y tres observados exclusivamente en el género Coffea 12,33. Estos motivos están relacionados tanto con la función catalítica, como con las regiones de unión con los otros dominios proteicos 34. El dominio CC de los CNL-CA presentó tres motivos altamente conservados identificados exclusivamente en secuencias del género Coffea, y patrones de motivos como los observados en los CNL de otras plantas. También pudimos identificar el motivo EDVL, un motivo similar a EDVID que se ha descrito en la mayoría de las proteínas CNL y que media la interacción molecular entre los dominios de los CNL. Tambien fue possible identificar otro motivos como P-loop y MHDL, los cuales estan relacionados con la unión a nucleótidos y la activación de proteínas. 35. Por otro lado, nuestras anotaciones de proteínas CNL-CA corresponden a estructuras de proteínas NBS-LRR típicas, lo que respalda la evidencia previa relacionada con la función de estas en la respuesta inmune de Coffea arabica 6,8,35.
Los análisis filogenéticos mostraron una distribución de CNL-CA similar a la observada en la soja y Arabidopsis, que agrupan CNL en cuatro clados filogenéticos principales. Nuestro árbol filogenético mostró cuatro clados principales, similares a otras plantas, siete subclados, además de una secuencia segregada 32,36. Además, estos resultados son consistentes con el análisis filogenético que compara las secuencias CNL de referencia y las secuencias CNL-CA. Este análisis mostró muchas relaciones filogenéticas entre CNL-CA y CNL de referencia de A. thaliana y la familia Solanaceae. El estudio realizado por Noir (2001) 12 demostró que el mayor número de genes de resistencia en Coffea arabica tiene una relación evolutiva con los genes de resistencia de tomate y Arabidopsis, similar a lo observado en este estudio. Los resultados filogeneticos de las secuencias Ca_3_358_ORF_2_1 y Ca_50_488_ORF2_1 sugieren que las estas pueden ser una respuesta adaptativa a un patógeno específico, lo que puede indicar que se trata de genes exclusivos del género Coffea. Con respect a la distribución cromosómica de los CNL-CA no fue uniforme, siendo este resultado similar a los reportes anteriores para C. arabica y otras especies de plantas 10,13,25,32,37. Además, el 71 % de los CNL-CA muestran distribuciones de copias en tándem en loci alineados con pseudocromosomas de C. canephora. Estos resultados son similares a los observados en otros estudios que relacionan estas distribuciones de genes con los procesos de adaptación de los patógenos de plantas 10,32,37. Además, estas distribuciones de genes podrían estar relacionadas con la susceptibilidad de NBS-LRR a los procesos de recombinación y duplicación genética. Se cree que estos fenómenos son el resultado de mecanismos coevolutivos planta-patógeno; que afectan la inmunidad de las plantas, especialmente en la inmunidad activada por efectores (ETI), donde NBS-LRR juega un papel relevante 26. Finalmente, las estructuras proteicas predichas de los CNL-CA presentaron homología con las proteínas de referencia con funciones similares, lo que confirma que los genes CNL-CA codifican proteínas que son estructuralmente similares a otras CNL vegetales previamente clonadas y dilucidadas 5,9.
CONCLUSIONES
Los resultados de este estudio sugieren que Coffea arabica tiene un número limitado de genes NBS-LRR en comparación con otras especies de café, lo que podría explicar su alta susceptibilidad a patógenos biotróficos. Sin embargo, la identificación de motivos altamente conservados observados exclusivamente en secuencias del género Coffea; la distribución cromosómica con copias en tándem de estos genes, podría indicar que son el resultado de la adaptación a patógenos exclusivos del café. Utilizar esta información sobre estos R-genes en programas de mejoramiento genético del café podria ser una estrategia util para el Desarrollo de nuevas variedades de café, principalmente en el caso variedades resistentes a patógenos.
Materiales Supplementarios:
Tabla Suplementaria 1: Anotaciones de secuencias
Tabla Suplementaria 2: Localización cromosómica.
Author Contributions: AO conceptualizó el estudio; MM, ME y JL contribuyeron con el diseño del estudio; AO, MM, ME realizó el análisis in silico. Todos los autores escribieron, revisaron, leyeron y aprobaron el manuscrito.
Funding: Este estudio fue financiado por el Instituto de Investigaciones en Microbiología y el Instituto Hondureño del Café.
Agradeciminetos: Los autores agradecen el apoyo brindado por el personal del instituto de investigación en microbiología y del Instituto Hondureño del Café, en particular Yonis Morales, Cristian Lizardo y Diana Herrera. Agradecemos la ayuda brindada por Gustavo Fontecha y Gabriela Matamoros en la revisión del manuscrito final, así como el apoyo del profesor Flavio Henrique Silva de la Universidad Federal de Sao Carlo, por su orientación para el desarrollo de este enfoque.
Conflictos de Intereses: Los autores declaran no tener conflicto de intereses. Los financiadores no tuvieron ningún papel en el diseño del estudio; en la recopilación, análisis o interpretación de datos; en la redacción del manuscrito, o en la decisión de publicar los resultados.
REFERENCCIAS
1 Lashermes P, Combes MC, Robert J, Trouslot P, D’Hont A, Anthony F et al. Molecular characterisation and origin of the Coffea arabica L. Genome. Molecular and General Genetics 1999; 261: 259–266.
2 Talhinhas P, Batista D, Diniz I, Vieira A, Silva DN, Loureiro A et al. The coffee leaf rust pathogen Hemileia vastatrix: one and a half centuries around the tropics. Molecular Plant Pathology 2017; 18: 1039–1051.
3 Jones, J.D.G. DanglJL. H E P L a N T Imm U N E S Y S T. Nature 2006; 444: 323–329.
4 Dangl JL, Jones JDG. Dangl_pathogen_defense_review_2001_nature. 2001; 411.
5 McHale L, Tan X, Koehl P, Michelmore RW. Plant NBS-LRR proteins: Adaptable guards. Genome Biology 2006; 7. doi:10.1186/gb-2006-7-4-212.
6 Ausubel FM. Are innate immune signaling pathways in plants and animals conserved? Nature Immunology 2005; 6: 973–979.
7 Williams SJ, Sohn KH, Wan L, Bernoux M, Sarris PF, Segonzac C et al. Structural basis for assembly and function of a heterodimeric plant immune receptor. Science (1979) 2014; 344: 299–303.
8 Dubey N, Singh K. Role of NBS-LRR proteins in plant defense. Molecular Aspects of Plant-Pathogen Interaction 2018; : 115–138.
9 Bella J, Hindle KL, McEwan PA, Lovell SC. The leucine-rich repeat structure. Cellular and Molecular Life Sciences 2008; 65: 2307–2333.
10 Cheng X, Jiang H, Zhao Y, Qian Y, Zhu S, Cheng B. A genomic analysis of disease-resistance genes encoding nucleotide binding sites in Sorghum bicolor. Genetics and Molecular Biology 2010; 33: 292–297.
11 Yang S, Zhang X, Yue JX, Tian D, Chen JQ. Recent duplications dominate NBS-encoding gene expansion in two woody species. Molecular Genetics and Genomics 2008; 280: 187–198.
12 Noir S, Combes MC, Anthony F, Lashermes P. Origin, diversity and evolution of NBS-type disease-resistance gene homologues in coffee trees (Coffea L.). Molecular Genetics and Genomics 2001; 265: 654–662.
13 Alvarenga SM, Caixeta ET, Hufnagel B, Thiebaut F, Maciel-Zambolim E, Zambolim L et al. In silico identification of coffee genome expressed sequences potentially associated with resistance to diseases. Genetics and Molecular Biology 2010; 33: 795–806.
14 Rodrigues CJ, Bettencourt AJ, Rijo L. Races of the Pathogen and Resistance to Coffee Rust. Annual Review of Phytopathology 1975; 13: 49–70.
15 Sera GH, Sera T, Ito DS, de Azevedo JA, da Mata JS, Dói DS et al. Resistance to leaf rust in coffee carrying SH3 gene and others SH genes. Brazilian Archives of Biology and Technology 2007; 50: 753–757.
16 Sekhwal MK, Li P, Lam I, Wang X, Cloutier S, You FM. Disease resistance gene analogs (RGAs) in plants. International Journal of Molecular Sciences 2015; 16: 19248–19290.
17 Osuna-Cruz CM, Paytuvi-Gallart A, di Donato A, Sundesha V, Andolfo G, Cigliano RA et al. PRGdb 3.0: A comprehensive platform for prediction and analysis of plant disease resistance genes. Nucleic Acids Research 2018; 46: D1197–D1201.
18 Finn RD, Coggill P, Eberhardt RY, Eddy SR, Mistry J, Mitchell AL et al. The Pfam protein families database: Towards a more sustainable future. Nucleic Acids Research 2016; 44: D279–D285.
19 Camacho C, Coulouris G, Avagyan V, Ma N, Papadopoulos J, Bealer K et al. BLAST+: Architecture and applications. BMC Bioinformatics 2009; 10: 1–9.
20 Artimo P, Jonnalagedda M, Arnold K, Baratin D, Csardi G, de Castro E et al. ExPASy: SIB bioinformatics resource portal. Nucleic Acids Research 2012; 40: 597–603.
21 Jones P, Binns D, Chang HY, Fraser M, Li W, McAnulla C et al. InterProScan 5: Genome-scale protein function classification. Bioinformatics 2014; 30: 1236–1240.
22 Bailey TL, Boden M, Buske FA, Frith M, Grant CE, Clementi L et al. MEME Suite: Tools for motif discovery and searching. Nucleic Acids Research 2009; 37: 202–208.
23 Dereeper A, Bocs S, Rouard M, Guignon V, Ravel S, Tranchant-Dubreuil C et al. The coffee genome hub: A resource for coffee genomes. Nucleic Acids Research 2015; 43: D1028–D1035.
24 Kelley LA, Mezulis S, Yates CM, Wass MN, Sternberg MJE. The Phyre2 web portal for protein modeling, prediction and analysis. Nature Protocols 2015; 10: 845–858.
25 Jacob F, Vernaldi S, Maekawa T. Evolution and conservation of plant NLR functions. Frontiers in Immunology 2013; 4: 1–16.
26 Qian LH, Zhou GC, Sun XQ, Lei Z, Zhang YM, Xue JY et al. Distinct patterns of gene gain and loss: Diverse evolutionary modes of NBS-encoding genes in three solanaceae crop species. G3: Genes, Genomes, Genetics 2017; 7: 1577–1585.
27 Zhang X, Feng Y, Cheng H, Tian D, Yang S, Chen JQ. Relative evolutionary rates of NBS-encoding R-genesevealed by soybean segmental duplication. Mol Genet Genomics 2011; 285: 79–90.
28 Jupe F, Pritchard L, Etherington GJ, MacKenzie K, Cock PJA, Wright F et al. Identification and localisation of the NB-LRR gene family within the potato genome. BMC Genomics 2012; 13: 1–14.
29 Shao ZQ, Zhang YM, Hang YY, Xue JY, Zhou GC, Wu P et al. Long-term evolution of nucleotide-binding site-leucine-rich repeat genes: Understanding gained from and beyond the legume family. Plant Physiology 2014; 166: 217–234.
30 Meyers BC, Morgante M, Michelmore RW. TIR-X and TIR-NBS proteins: Two new families related to disease resistance TIR-NBS-LRR proteins encoded in Arabidopsis and other plant genomes. Plant Journal 2002; 32: 77–92.
31 Zhang YM, Shao ZQ, Wang Q, Hang YY, Xue JY, Wang B et al. Uncovering the dynamic evolution of nucleotide-binding site-leucine-rich repeat (NBS-LRR) genes in Brassicaceae. Journal of Integrative Plant Biology 2016; 58: 165–177.
32 Wei H, Liu J, Guo Q, Pan L, Chai S, Cheng Y et al. Genomic Organization and Comparative Phylogenic Analysis of NBS-LRR Resistance Gene Family in Solanum pimpinellifolium and Arabidopsis thaliana. Evolutionary Bioinformatics 2020; 16. doi:10.1177/1176934320911055.
33 Hendre PS, Bhat PR, Krishnakumar V, Aggarwal RK, Donini P. Isolation and characterization of resistance gene analogues from Psilanthus species that represent wild relatives of cultivated coffee endemic to India. Genome 2011; 54: 377–390.
34 Xue JY, Zhao T, Liu Y, Liu Y, Zhang YX, Zhang GQ et al. Genome- Wide Analysis of the Nucleotide Binding Site Leucine-Rich Repeat Genes of Four Orchids Revealed Extremely Low Numbers of Disease Resistance Genes. Frontiers in Genetics 2020; 10: 1–12.
35 Rairdan GJ, Collier SM, Sacco MA, Baldwin TT, Boettrich T, Moffett P. The coiled-coil and nucleotide binding domains of the potato Rx disease resistance protein function in pathogen recognition and signaling. Plant Cell 2008; 20: 739–751.
36 Nepal MP, Benson B v. CNL disease resistance genes in soybean and their evolutionary divergence. Evolutionary Bioinformatics 2015; 11: 49–63.
37 Zhao Y, Weng Q, Song J, Ma H, Yuan J, Dong Z et al. Bioinformatics Analysis of NBS-LRR Encoding Resistance Genes in Setaria italica. Biochemical Genetics 2016; 54: 232–248.
Received: 21 March 2022 / Accepted: 27 july 2022 / Published:15 August 2022
Citation: Moncada M M, Elvir M A, Lopez J R, Ortiz A S. Predicción bioinformática de proteínas NBS-LRR en el genoma de Coffea arabica. Revis Bionatura 2022;7(3) 19. http://dx.doi.org/10.21931/RB/2022.07.03.19



















