Vol 6 No 1 2021 – 13
INVESTIGATION / RESEARCH
Evaluación de la diversidad genética de una colección venezolana de morera mediante marcadores moleculares.
Evaluation of the genetic diversity of a Venezuelan collection of mulberry using molecular markers
Ramón Rea1, Sandy Molina-Moret1, Darío Torrealba1Sabino Jiménez1, Laura Gelves1 y Daynet Sosa2
Available from: http://dx.doi.org/10.21931/RB/2021.06.01.13
RESUMEN
La morera, Morus alba L. es un árbol leñoso, perenne de porte bajo a medio y de amplia adaptabilidad, tanto en áreas templadas como tropicales. Sus principales usos son en sericultura, alimentación animal y humana. El presente trabajo evaluó la diversidad genética de una colección de 28 entradas de morera usando marcadores moleculares ISSR. Los cinco iniciadores ISSR utilizados para la amplificación produjeron un total de 115 fragmentos polimórficos, con un tamaño entre 100-1800 pares de bases (pb), permitiendo la separación de cuatro grupos principales de diversidad, utilizando la disimilaridad debida al coeficiente de Dice y Análisis de Coordenadas Principales (ACoP). El primer grupo formado por las accesiones: Boconó-6, Maracay-1, Maracay-2, Maracay-3, Táchira-3, Táchira-4, Táchira-5 y Táchira-6; el segundo grupo por Boconó-3, Española-1, Murcia-1, Murcia-2, Murcia-3 y Yu-2; el tercer grupo por Boconó-1, Boconó-2, Maracay-6, Táchira-1, Táchira-2 y Yu-62 y el cuarto grupo por ACC-1, ACC-2, ACC-3, CGT-1, CGT-2, CGT-3, Española-3 y Española-7, con una diversidad genética de 0.242, 0.169, 0.230 y 0.219, respectivamente. Estos resultados constituyen una aproximación al conocimiento de la diversidad genética del cultivo en nuestro país, con fines de iniciar los programas de pre-mejora y mejora genética.
Palabras clave: ISSR, Morus alba L., sericultura, similitud genética
ABSTRACT
Mulberry, Morus alba L., is a woody, perennial tree of small to medium size and broad adaptability in temperate and tropical areas. Its main uses are in sericulture, animal, and human feeding. This work studied the genetic diversity of a Venezuelan collection of Mulberry by ISSR molecular markers. The five ISSR primers used for amplification produced 115 polymorphic fragments, with a size between 100-1800 base pairs (bp), allowing the separation of four major diversity groups using dissimilarity to the Dice coefficient and Principal Coordinates Analysis (PCoA). The first group formed by the accessions: Boconó-6, Maracay-1, Maracay-2, Maracay-3, Táchira-3, Táchira-4, 5-Táchira, Táchira-6; the second group by Boconó-3, Española-1, Murcia-1, Murcia-2, Murcia-3 and Yu-2; the third group by Boconó-1, Boconó-2, Maracay-6, Táchira-1, Táchira-2 and Yu-62 and the fourth group by ACC-1, ACC-2, ACC-3, CGT-1, CGT-2, CGT-3, Española-3 and Española-7, with genetic diversity of 0.242, 0.169, 0.230 and 0.219, respectively. These results provide a better knowledge of mulberry’s genetic diversity in our country to initiate pre-breeding and breeding programs.
Keywords: ISSR, Morus alba L., sericulture, genetic similarity
INTRODUCCIÓN
La morera (Morus alba L.) es un árbol leñoso, perenne de porte bajo a medio y de amplia adaptabilidad, tanto de áreas templadas como tropicales en Asia, Europa, Norte América, Sur América y África. La morera, género Morus, pertenece a la familia Moraceae, de las cuales hay al menos 24 especies conocidas1. La morera es usada como única fuente de alimento para el gusano de seda Bombix mori en la sericultura. El follaje también es usado como alimento animal por su alto contenido nutritivo. En las regiones tropicales, la predisposición a utilizar el follaje de especies arbustivas para la producción animal ha estado vigente en los últimos años, y cobra importancia en respuesta al potencial de producción de nutrientes en las arbóreas, que se relaciona con altos niveles de proteína cruda (>18 %), alta digestibilidad, excelente balance de minerales y por la capacidad de producir forraje aún en época de sequía2. En este sentido, trabajos recientes sugieren el uso del follaje de plantas arbustivas con potencial forrajero, como complemento alimenticio para corregir las carencias de nutrientes que presentan los pastos en las regiones tropicales. Una iniciativa para muchos países tropicales podría ser también la siembra densa de Morera para utilizarla como forraje3. Su uso como alimento comenzó a partir de la década del ochenta en América Central debido a sus excelentes características bromatológicas4.En general Morus alba L., es una planta multifuncional con propiedades farmacológicas, con flavonoides como constituyentes principales, las hojas de morera poseen diversas actividades biológicas, que incluyen actividades antioxidantes, antimicrobianas, blanqueadoras de la piel, citotóxicas, antidiabéticas, inhibidoras de glucosidasa, antihiperlipidémicas, antiateroscleróticas, antiobesidad, cardioprotectoras y de mejora cognitiva. Ricas en antocianinas y alcaloides, las frutas de morera tienen propiedades farmacológicas, como actividades antioxidantes, antidiabéticas, antiateroscleróticas, antiobesidad y hepatoprotectoras. La corteza de la raíz de la morera, que contiene flavonoides, alcaloides y estilbenoides, tiene propiedades antimicrobianas, blanqueadoras de la piel, citotóxicas, antiinflamatorias y antihiperlipidémicas. El consumo de preparaciones de las hojas de morera controla comorbilidades relacionadas con la complejidad de la COVID-19 como la Diabetes mellitus tipo 1 y 2 y las enfermedades cardiovasculares5, 6. Todo esto adquiere relevancia para observar la evolución hacia cuadros complejos presentes en la severidad de la COVID-19, relacionada con una respuesta disfuncional del sistema inmune7, 8.
La evaluación del nivel y distribución de la diversidad genética dentro del germoplasma existente de morera, no solo puede contribuir al conocimiento sobre su historia evolutiva, sino que es fundamental para su conservación y mejora9. Se han desarrollados estudios previos sobre diversidad genética usando caracteres morfológicos y técnicas moleculares. Los marcadores moleculares son complementos útiles a los caracteres morfológicos y fenológicos debido a que son abundantes, independientes del tejido o de efectos ambientales y permiten identificar genotipos en estados iniciales de desarrollo de la planta. Estas técnicas revelan polimorfismos a nivel del ADN y son herramientas muy poderosas para la caracterización y estimación de la diversidad genética. En Morus sp., se han usado exitosamente estos marcadores moleculares con fines de caracterización tanto de cultivares como de especies silvestres, así como de análisis de diversidad genética. Entre los marcadores moleculares empleados se tiene los RAPD-PCR (reacción en cadena de la polimerasa para la amplificación al azar del ADN polimórfico)10,11 , AFLP (polimorfismos de longitud de fragmentos amplificados)12 , SSR (secuencias simples repetidas)13,14, siendo los ISSR (secuencias simples repetidas entre secuencias no repetitivas) los marcadores más comúnmente utilizados en Morus sp., con diferentes propósitos, siendo los más destacados, la identificación de especies e incluso variedades intra-especie, así como también los estudios de diversidad y estabilidad genética15,16,17
En Venezuela, se ha iniciado un programa de aprovechamiento integral de la morera en diferentes áreas agroecológicas con fines de alimentación animal, humana y sericultura. La Fundación Instituto de Estudios Avanzados (IDEA) ha recibido del Instituto Nacional de Investigaciones Científicas (IVIC) material colectado de siembra de diferentes localidades del país, desconociendo las relaciones genéticas entre ellas. En tal sentido, el objetivo de este trabajo fue evaluar la variabilidad genética de genotipos de morera colectados en Venezuela mediante marcadores ISSR.
MATERIALES Y MÉTODOS
Material vegetal, extracción y amplificación del ADN
Se tomaron muestras de lo que inicialmente se consideraban 8 diferentes variedades de morera, cuyo origen no se conoce exactamente y se realizó la extracción de ADN de hojas jóvenes mediante el protocolo de Dellaporta18.
La cantidad y pureza del ADN extraído se midió en un espectrofotómetro Eppendorf BioPhotometer mediante la determinación de la relación de absorbancias 260/280 nm, y su integridad se evaluó mediante electroforesis en geles de agarosa al 1% en tampón TAE 1X19. El ADN fue diluido a 1 ng/µL para las reacciones de amplificación de ISSR.
El análisis con los marcadores ISSR incluyó el uso de 5 iniciadores de 17-19 nucleótidos de longitud, desarrollados por el laboratorio de Biotecnología de la Universidad de la Columbia Británica (Tabla 1).

*Abreviaciones para posiciones de bases mezcladas: H= no G; V=no T.
Tabla 1. Lista de iniciadores de tipo ISSR seleccionados para la amplificación y evaluación de variedades de morera.
Para las reacciones de amplificación de ISSR-PCR se utilizaron: tampón de PCR 1X, 3 mM MgCl2, 0.2 mM dNTPs (Promega Co. Ltd.), formamida al 2%, 0.25 µM del iniciador en estudio, 2 U Taq polimerasa (Promega Co. Ltd.) y 2.5 ng de ADN genómico en un volumen final de 20 mL. Las condiciones de amplificación fueron las siguientes: (Tabla 2).

Tabla 2. Condiciones de amplificación de fragmentos ISSR utilizada.
Todas las reacciones se realizaron en un termociclador Cycler (Bio-Rad). Los productos amplificados se analizaron mediante electroforesis en geles de agarosa al 1,5% en tampón TAE 1X, mediante comparación con un marcador de peso molecular (1kb plus (Invitrogen) y fueron visualizados mediante un escáner Typhoon (GE Healthcare). La interpretación genética de los patrones de ISSR, esencialmente de naturaleza dominante, se basó en que solamente existen dos fenotipos, los que presentan la banda que son los individuos homocigotos dominantes y los heterocigotos y los que no la presentan que son los individuos homocigotos recesivos.
La nomenclatura utilizada consistió en señalar cada banda con el nombre y el número del iniciador comercial seguido de otro número que se refiere a la posición de la banda en el orden de menor a mayor migración en los geles.
Análisis de Datos
En ausencia de análisis de segregación no se hizo ningún supuesto sobre la naturaleza genética de los alelos, los fragmentos de amplificación fueron codificados de acuerdo a un marcador dominante, es decir, A1A1=A1A2=1 y A2A2=0, generando una columna por locus para cada iniciador. El nivel de polimorfismo y la capacidad discriminatoria de cada iniciador se valoró a través el contenido de información polimórfica (PIC) y la probabilidad de obtener parejas idénticas de alelos entre las muestras estudiadas20. La colección venezolana de morera consta de nueve genotipos y de allí se tomaron 29 individuos; cuatro de Bocono, cuatro de Maracay, seis de Táchira, tres Española, tres Murcia, tres ACC, tres CGT, una Yu-2 y una YU-62. La relación genética entre los nueve genotipos fue estudiada aplicando el uso combinado del Análisis de Coordenadas Principales (ACoP) y el Análisis de Conglomerados (AC), sobre datos de disimilitud utilizando los coeficientes de Jaccard, Emparejamiento simple, Dice y Rogers y Tanimoto20. El número k de dimensiones a ser retenidas, el coeficiente de similitud que mejor define la estructura de los datos y las medidas de la calidad fueron calculados utilizando también los procedimientos descritos por Demey20. Todos los análisis se realizaron utilizando InfoStat 2013 21 e Info-Gen 201122
RESULTADOS Y DISCUSIÓN
Los cinco iniciadores ISSR utilizados para la amplificación produjeron un total de 134 fragmentos polimórficos, con un tamaño entre 100-1800 pares de bases (pb). Tamaños similares han sido descritos por otros autores quienes describen bandas amplificadas comprendidas entre 520-2200pb23 y entre 200 – 2000pb1.
El iniciador ISSR-815 fue el que generó el menor número de fragmentos amplificados (13 bandas) y el mayor número de fragmentos fue generado con el iniciador ISSR-823 (37 bandas). Otros autores, con el empleo de estos marcadores, han obtenido altos niveles de polimorfismo para Morus sp. Uno de los trabajos reporta que estudiando las relaciones genéticas entre 21 genotipos de morera y utilizando 11 iniciadores ISSR se obtuvo un 96.55% de bandas polimórficas 23. En otro estudio donde se evaluaron 20 genotipos con 8 iniciadores ISSR obtuvieron un 97.6 % de polimorfismo24. Otro estudio donde utilizaron 11 ISSR obtuvieron un promedio de 91.78% de marcadores polimórficos1. Sin embargo, también se han descrito niveles más bajos de polimorfismo para este género, aun cuando se plantea que existe más variabilidad genética entre genotipos de M. alba en contraste con la encontrada en otras especies como M. nigra 12,25. Así tenemos el polimorfismo de 91.3% descrito en 27 variedades de cultivo y silvestres de Morera con el empleo de 15 marcadores ISSR14; en genotipos colectados de diferentes países encuentran un 74.13% de polimorfismo con el empleo de 10 ISSR26; otro estudio encuentra solo un 50% de polimorfismo entre 24 cultivares con el empleo de 17 marcadores13.
Los valores del Contenido de Información Polimórfica (PIC) para cada marcador ISSR variaron de 0.16 a 0.29; el marcador ISSR-891 fue el iniciador más informativo del conjunto, con un valor PIC mayor al 50% del intervalo teórico de 0.01 a 0.50 y además mostró la mayor capacidad discriminatoria, evaluada a través de la probabilidad de que dos variedades diferentes tengan igual identidad (Tabla 3).

*Probabilidad de que dos individuos para el Loci evaluado tengan igual identidad
Tabla 3. Nivel de polimorfismo y capacidad discriminatoria de los iniciadores
En la figura 1, se muestra el espacio bidimensional obtenido del Análisis de Coordenadas Principales (ACoP), utilizando el coeficiente de similitud de Dice, que según la metodología de análisis propuesta, refleja la mayor coherencia entre la matriz de distancias observadas y estimadas, lo cual garantiza la mayor estabilidad de la representación y ofrece una mejor aproximación de las relaciones entre las variedades. Las dos primeras dimensiones explican el 20% de la variabilidad total y permiten la formación de cuatro grupos de individuos utilizando los cinco iniciadores ISSR. El primer grupo formado por los individuos Boconó-6, Maracay-1, Maracay-2, Maracay-3, Táchira-3, Táchira-4, Táchira-5 y Táchira-6; el segundo grupo por Boconó-3, Española-1, Murcia-1, Murcia-2, Murcia-3 y Yu-2; el tercer grupo por Boconó-1, Boconó-2, Maracay-6, Táchira-1, Táchira-2 y Yu-62; y el cuarto grupo por ACC-1, ACC-2, ACC-3, CGT-1, CGT-2, CGT-3, Española-3 y Española-7, con una diversidad genética de 0.242, 0.169, 0.230 y 0.219, para el primero, segundo, tercer y cuarto grupo, respectivamente, lo cual ocurre con frecuencia ya que usualmente se utilizan especies seleccionadas por características similares de rendimiento y adaptación al ambiente. En términos de similitud, los resultados permiten probar que dos individuos con posiciones más contiguas en la representación bidimensional, tendrán patrones más similares de ADN respecto a los iniciadores utilizados.
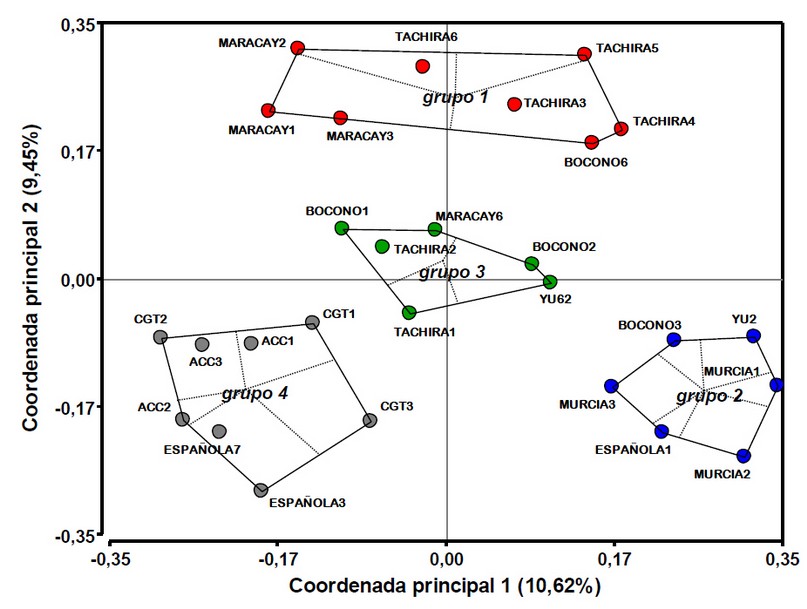
Figura 1. Relaciones genéticas entre los 28 individuos de la colección venezolana de morera basada en la disimilaridad debida al coeficiente de Dice y los cinco iniciadores ISSR.
Basados en los resultados obtenidos, no se puede afirmar que exista un patrón de agrupamiento debido a la procedencia de los individuos estudiados, similar a lo encontrado en el banco de germoplasma de Morera de la granja experimental “El Pílamo” de la Universidad Tecnológica de Pereira (Colombia), donde encontraron altos valores en los aspectos de estadística descriptiva y diferenciación poblacional que demuestran la gran complejidad presente en el genoma de morera17. Esto podría estar indicando el entrecruzamiento inducido por parte de los Fito mejoradores de variedades geográficamente distantes, lo que genera un acercamiento desde el punto de vista del material genético de los individuos.
A pesar de la similitud entre grupos, se nota que en el grupo 4 formado a partir de los ACoP, existe la predominancia de individuos de las variedades ACC y CGT, los cuales están ausentes en los demás grupos, por lo que se podría indicar que precisamente estas variedades, que en principio se creían distintas, pertenecen en realidad a un mismo grupo varietal de M. alba L. El conocimiento de las relaciones genéticas del material de partida es necesario para garantizar el uso adecuado del genoma de una especie cuando se pretende aplicar procesos de hibridación y selección en un programa de mejora genética.
CONCLUSIONES
Los cinco marcadores ISSR evaluados no permitieron generar diferencias en el grupo genético debido a la procedencia de los individuos estudiados, lo que indica una baja variabilidad genética de morera en Venezuela. Es necesaria la inclusión en el estudio de nuevos marcadores ISSR y más genotipos para tener una mejor imagen de la estructura genética del germoplasma de M. alba L en Venezuela. Lo otro sería, la introducción de material foráneo de morera para aumentar la diversidad genética para futuros programas de mejora genética
AGRADECIMIENTOS
Al Instituto de Estudios Avanzados (IDEA) y al Fondo Nacional de Ciencia y Tecnología (FONACIT) por el apoyo y financiamiento para la ejecución de este trabajo.
REFERENCIAS
1. Choudhary, R., R. Chaudhury, S. K. Malik, S. Kumar y D. Pal. 2013. Genetic stability of mulberry germplasm after cryopreservation by two-step freezing technique. Afr. J. Biotechnol. 12 (41): 5983-5993.
2. Hove L., J.H Topps, S. Sibanda, L.R. Ndlovu. 2001. Nutrient intake and utilization by goats fed dried leaves of the shrub legumes Acacia angustissima, Calliandra colothyrsus and Leucaena leucocephala as supplements to native pasture hay. Anim. Feed Sci. Tech. 91:95-106.
3. Makkar, H. 2014. Aumento sostenible de la productividad del ganado mediante la utilización eficiente de los recursos alimenticios en países en vías de desarrollo. Revista Cubana de Ciencia Agrícola, 48(1): 55-58.
4. Medina, M. G., D. E. García, T. Clavero, J. Iglesias y J.G. López. 2007. Evaluación inicial de la morera (Morus alba L.) en condiciones de vivero. Zootecnia Tropi. 25(1):43-49.
5. Lown, Mark; Fuller, Richard; Lightowler,Helen; Fraser,Ann; Gallagher, Andrew; Stuart, Beth; Byrne, Christopher; Lewith, George. 2017. «Mulberry-extract improves glucose tolerance and decreases insulin concentrations in normoglycaemic adults : Results of a randomised double-blind placebo-controlled study».:1-14.
http://dx.doi.org/10.1371/journal.pone.0172239.
6. Thaipitakwong Thanchanit, Surawej Numhom, y Pornanong Aramwit. (2018). «Mulberry leaves and their potential effects against cardiometabolic risks: A review of chemical compositions, biological properties and clinical efficacy». Pharmaceutical Biology 56(1): 109-18
7. Wei-jie Guan, Zheng-yi Ni, Yu Hu, Wen-hua Liang, Chun-quan Ou, Jian-xing He, Lei Liu, Hong Shan, Chun-liang Lei, David, Hui, Bin Du, Lan-juan Li, et al. (2020). «Clinical Characteristics of Coronavirus Disease 2019 in China». New England Journal of Medicine: 1-13.
8. Kwong, Paul C, y Yuan-chi Lin. (2020). «A strategy of traditional Chinese medicine against COVID-19: linking current basic research and ancient medicine texts». 13(2): 79-81.
9. Banerjee, R., Chattopadhyay, S., y A. K. Saha (2016). Genetic diversity and relationship of mulberry genotypes revealed by RAPD and ISSR markers, Journal of Crop Improvement, DOI: 10.1080/15427528.2016.1177803.
10. Zhao, W.G. y Y. Pan. (2004). Genetic Diversity of Genus Morus Revealed by RAPD Markers. Int. J. Agri. Biol., 6(6):950-955.
11. Manjula, A. C., Kote, N. V., Shivashankarappa, L. H., Vishwanatha, T., y Patil, A. G. (2020). Evaluation of genetic diversity amongst mulberry varieties Morus alba by RAPD marker. Plant Cell Biotechnology and Molecular Biology, 21(45-46), 138-145. Retrieved from https://ikprress.org/index.php/PCBMB/article/view/5510
12. Kafkas, S., M. Özgen, Y. Doğan, B. Özcan, S. Ercişli y S. Serçe. 2008. Molecular characterization of mulberry accessions in Turkey by AFLP markers. J. Amer. Soc. Hort. Sci. 133 (4): 593–597.
13. Zhao, W. G., X. X. Miao, B. Zang, L. Zhang, Y. L. Pan y Y. P. Huang. 2006. Construction of Fingerprinting and Genetic Diversity of Mulberry Cultivars in China by ISSR Markers. Acta Genetica Sinica. 33 (9): 851–860.
14. Zhao, W. G., Z. H. Zhou, X. X. Miao, Y. Zhang, S. B. Wang, J. H. Huang, H. Xiang, Y. L. Pan y Y. P. Huang. 2007. A comparison of genetic variation among wild and cultivated Morus species (Moraceae: Morus) as revealed by ISSR and SSR markers. Biodivers. Conserv. 16 (2): 275–290.
15. Awasthi, A. K., G. M. Nagaraja, G. V., Naik, S., Kanginakudru, K., Thangavelu y J. Nagaraju. 2004. Genetic diversity and relationships in mulberry (genus Morus) as revealed by RAPD and ISSR marker assays. BMC Genetics 5: 1-9.
16. Vijayan, K., A. Tikader, P. K. Kar, P. P. Srivastava, A. K. Awasthi, K. Thangavelu, B. Saratchandra. 2006. Assessment of genetic relationships between wild and cultivated mulberry (Morus) species using PCR based markers. Genet. Resour. Crop Evol. 53 (5): 873–882.
17. Gaviria-Arias, D., E. Aguilar-Fernández, H., Navia-Morocho y A. Alegría-Soto. 2012. Diversidad genética en el banco de germoplasma de morera [Morus spp. (Rosales: Moráceas)] de la granja experimental “el Pilamo”, Universidad Tecnológica de Pereira, Pereira (Risaralda), Colombia. actual biol. 34 (96): 33-42.
18. Dellaporta, S. L., J. Wood y J. B. Hicks. 1983. A plant DNA minipreparation version II. Plant Mol. Biol. Rep. 1 (4): 61-64.
19. Maniatis, T., E. F. Fritsch, y J. Sambrook. 1982. Molecular cloning: a laboratory manual. Cold Spring Harbor Laboratory, Cold Spring Harbor, N.Y.
20. Demey, J. R., J. L. Vicente-Villardón, M. P. Galindo-Villardón y A. Y. Zambrano. 2008. Identifying molecular markers associated with classification of genotypes by External Logistic Biplots. Bioinformatics. 24 (24): 2832-2838.
21. Di Rienzo, J. A., F. Casanoves, M. G. Balzarini, L. Gonzalez, M. Tablada y C. W Robledo. InfoStat versión 2013. Grupo InfoStat, FCA, Universidad Nacional de Córdoba, Argentina. En: http://www.infostat.com.ar.
22. Balzarini, M. G. y J. A. Di Rienzo. InfoGen versión 2011. FCA, Universidad Nacional de Córdoba, Argentina. En: http://www.info-gen.com.ar.
23. Ipek, M., L. Pirlak y S. Kafkas. 2012. Molecular characterization of mulberry (Morus spp) genotypes via RAPD and ISSR. J. Sci. Food Agric. 92 (8): 1633-1637.
24. Chikkaswamy, B. K., R. C. Paramanik, A. Debnath y S. M. Sadana. 2012. Evaluation of Genetic Diversity in Mulberry Varieties Using Molecular Markers. Nat. Sci. 10 (6): 45-60.
25. Orhan, E. y S. Ercisli. 2010. Genetic relationships between selected Turkish mulberry genotypes (Morus spp) based on RAPD markers. Genet. Mol. Res. 9 (4): 2176-2183.
26. Srivastava, P. P., K. Vijayan, A. K. Awasthi y B. Saratchandra. 2004. Genetic analysis of Morus alba through RAPD and ISSR markers. IJBT. 3 (4): 527-532.
Recibido: 18 diciembre2020
Aceptado: 10 enero 2021
Ramón Rea1, Sandy Molina-Moret1, Darío Torrealba1Sabino Jiménez1, Laura Gelves1 y Daynet Sosa2
1Fundación Instituto de Estudios Avanzados (IDEA), Carretera Nacional Hoyo de la Puerta, Valle de Sartenejal, Baruta, estado Miranda, Caracas 1015-A, Apartado17603, Venezuela.
2Escuela Superior Politécnica del Litoral, ESPOL, Facultad de Ciencias Naturales y Matemáticas, Guayaquil, Ecuador.
Autor de correspondencia: ramonrea@hotmail.com



















